| |
| Med Sci (Paris). 33(11): 963–970. doi: 10.1051/medsci/20173311013.3’RR Docteur Jekyll et Mister Hyde de la lymphopoïèse/lymphomagenèse B Alexis Saintamand,1*a Nour Ghazzaui,1 Hussein Issaoui,1 and Yves Denizot1b 1UMR CNRS 7276, Université de Limoges, rue Pr Descottes, 87025Limoges, France |
Vignette (Photo © Bertrand Nadel). Les différents événements géniques qui s’effectuent lors de la lymphopoïèse B (établissement du répertoire antigénique via les recombinaisons V(D)J1, maturation d’affinité par le processus d’hypermutations somatiques, et modification des fonctions effectrices de l’immunoglobuline (Ig) par la recombinaison de classe) requièrent une régulation extrêmement fine, à la fois dans le temps et l’espace (
Figure 1
) [1]. Leur déroulement séquentiel, qui nécessite la génération de cassures doubles brins de l’ADN, est assuré par différents mécanismes permettant de modifier l’accessibilité des différentes régions cibles des recombinaisons : régulation de la transcription, condensation de la chromatine, localisation des locus au sein du noyau, etc. La coordination de ces mécanismes est assurée principalement par des éléments cis-régulateurs2, répartis tout au long des locus d’Ig, particulièrement au locus des chaînes lourdes (IgH) (
Figure 2
) [1]. Un activateur transcriptionnel (ou enhancer, selon la terminologie anglo-saxonne) est défini comme une séquence d’ADN capable de stimuler à distance la transcription d’un gène, notamment en servant de site de fixation à des facteurs de transcription. Le locus IgH murin en comprend deux principaux : l’activateur intronique Eμ, localisé entre les segments JH et le gène Cμ, et la région régulatrice située en 3’ du locus (3’RR) en aval du gène Cα. C’est cette dernière qui coordonne toutes les phases tardives de la maturation B que sont la transcription du locus IgH, l’hypermutation somatique et la recombinaison de classe, assurant ainsi le fonctionnement optimal du lymphocyte B. La région 3’RR peut également contrôler la transcription d’oncogènes transloqués au locus IgH à la faveur des cassures chromosomiques, ce qui aboutit au processus de lymphomagenèse. Voilà donc le double visage de la région 3’RR…
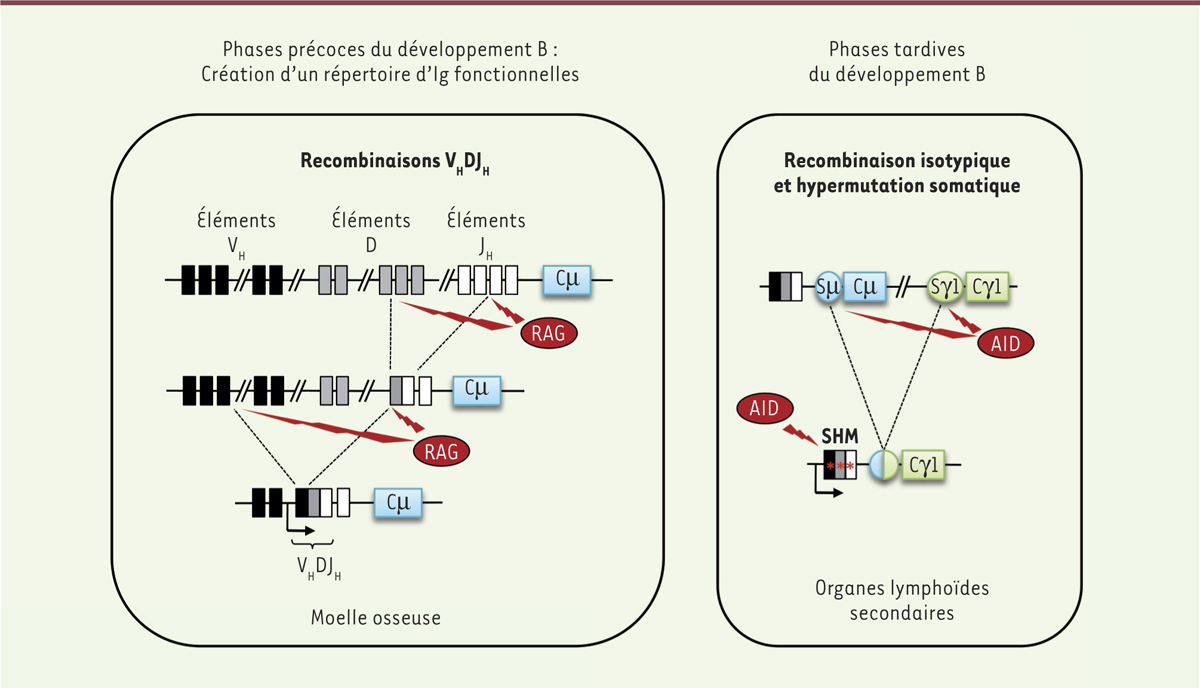
 | Figure 1.
Représentation schématique des événements de recombinaison au locus IgH lors de la lymphopoïèse B. Lors des phases précoces qui précèdent la rencontre avec l’antigène, les réarrangements VHDJH permettent, avec les réarrangements des chaines légères, l’expression d’une immunoglobuline (Ig) fonctionnelle. Les cassures chromosomiques créées pour ces réarrangements reposent sur l’activité de l’enzyme RAG (recombination-activating gene). Au cours des phases tardives du développement B, qui s’effectuent en présence de l’antigène, les phénomènes d’hypermutations somatiques et de recombinaison isotypique visent à optimiser l’affinité du récepteur pour l’antigène et à modifier l’isotype d’Ig exprimée. Ces processus nécessitent l’intervention de l’enzyme AID (activation-induced cytidine deaminase). SHM : somatic hypermutation.
|
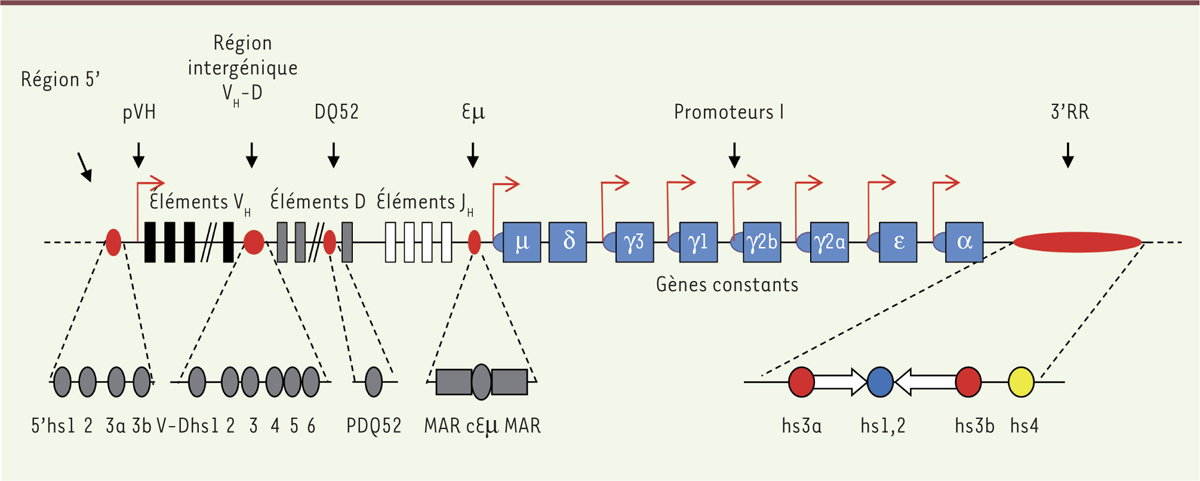
 | Figure 2.
Représentation schématique du locus IgH et de ses éléments cis-régulateurs chez la souris. La ligne supérieure représente le locus IgH murin en conformation germinale. Les éléments régulateurs sont représentés en rouge et sont détaillés sur la ligne inférieure. Les sites hs (pour hypersensibilité à la DNaseI) sont représentés par des cercles et des ovales, les MAR (matrix attachment regions) par des rectangles, et les séquences répétées inversées par des flèches blanches. hs3a et hs3b (cercles rouges de la ligne inférieure) possèdent 97 % d’homologies mais sont en orientation inversée. hs1,2 (cercle bleu) est l’élément central du palindrome constituant la région 3’RR. hs4 (cercle jaune) est situé en dehors du palindrome. Seuls Eµ et la 3’RR présentent une activité pro-transcriptionnelle importante. pVH : promoteur des régions variables. |
|
Eμ fut le premier activateur découvert au sein du locus IgH. Il est organisé sous forme d’un élément central (core Eμ ou cEμ) flanqué de deux régions d’attachement à la matrice nucléaire (MAR, pour matrix attachement region). La délétion d’Eμ perturbe fortement les réarrangements V(D)J au locus IgH mais n’a guère d’impact sur la transcription du locus, l’hypermutation somatique et la recombinaison de classe [2, 3]. D’autres éléments régulateurs pour le contrôle de ces processus ont donc été recherchés au sein du locus. Quatre nouveaux activateurs transcriptionnels, répartis dans une région régulatrice d’environ 30 kb, ont ainsi été identifiés en 3’ du gène constant Cα et nommés, de 5’ vers 3’, hs3a, hs1,2, hs3b et hs4 [1]. Le séquençage de cette région correspondant à la région 3’RR, a permis de mettre en évidence des caractéristiques structurales étonnantes. Une première particularité provient de son architecture palindromique qui couvre les trois premiers activateurs transcriptionnels, soit environ 25 kb (
Figure 3
) [4]. En effet, hs1,2 est flanqué de deux séquences répétées inversées (IR, pour inverted repeat), dont certaines sont en tandem (TR, pour tandem repeat) ; hs3a et hs3b forment les limites de ces séquences répétées inversées. Leurs séquences présentent 97 % d’homologie et ils sont disposés en orientation inverse, prolongeant ainsi le palindrome. L’élément hs4, quant à lui, se situe en dehors du palindrome et constitue la limite 3’ de la région 3’RR. La 3’RR serait divisée en deux unités distinctes : le palindrome et ses trois activateurs, hs1,2, hs3a et hs3b, d’une part, et hs4, d’autre part [5, 6]. Une étude plus fine, réalisée au laboratoire, a récemment révélé que les éléments transcriptionnels de la 3’RR étaient, d’autre part, flanqués de séquences répétitives similaires aux régions S (switch) qui sont essentielles à la recombinaison de classe [7, 8] (→). (→) Voir la Dernière heure de S. Péron et al., m/s n° 5, mai 2012, page 551
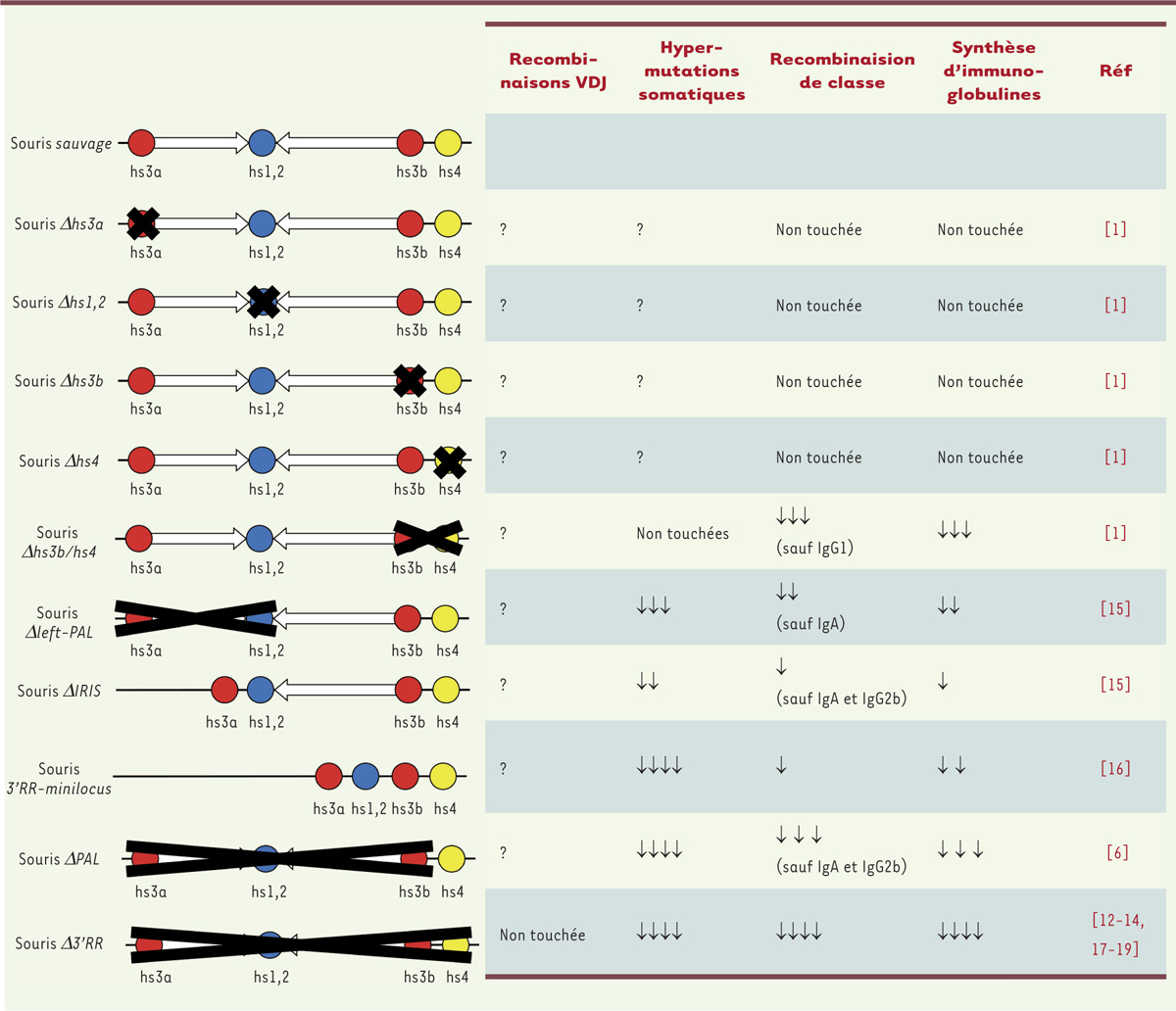
 | Figure 3.
Modèles murins de délétion de la région 3’RR. Différents modèles murins de délétions de la région 3’RR ont été générés et analysés. La délétion d’un seul activateur transcriptionnel n’a que peu ou pas d’effet, confirmant la redondance et la synergie d’activité entre les différents éléments de la 3’RR. La délétion de plusieurs activateurs perturbe fortement la recombinaison de classe, l’hypermutation somatique et la synthèse d’Ig. La suppression du palindrome a un effet notable sur l’hypermutation somatique. Cercles rouges : hs3a et hs3b ; cercles bleus : hs1,2 ; cercle jaune hs4 ; flèches blanches : régions répétées inversées du palindrome de la 3’RR. Le point d’interrogation (?) indique un paramètre non étudié. Le nombre de flèches vers le bas indique l’intensité de la baisse sur le paramètre étudié. |
De façon surprenante, ces séquences, baptisées séquences LS (pour like-switch) sont la cible de l’enzyme AID (activation-induced cytidine deaminase), ce qui permet une recombinaison similaire à la recombinaison de classe entre la séquence Sμ et la région 3’RR. Cette recombinaison, nommée LSR (pour locus suicide recombination), aboutit à la délétion de l’ensemble des gènes constants (C), ce qui conduit à l’apoptose des lymphocytes B matures qui ne peuvent survivre sans exprimer de BCR (B cell receptor) [7, 8]. Ce processus permettrait l’élimination des clones auto-réactifs. Chez l’homme, on retrouve également une région régulatrice en 3’ qui possède certaines particularités propres à la structure du locus IgH humain. En effet, les gènes constants y sont dupliqués et forment deux blocs distincts séparés d’environ 30 kb. Chaque bloc possède sa propre région 3’RR, localisée en aval des gènes Cα1 et Cα2. Chaque 3’RR ne contient que trois activateurs (hs1,2, hs3 et hs4) mais, comme chez la souris, présente une structure palindromique, hs1,2 étant flanqué de séquences répétées inversées [1, 9]. De manière intéressante, les homologies de séquences sont limitées entre espèces, tant pour les activateurs que pour les séquences IR, mais la structure palindromique de la région 3’RR est conservée chez tous les mammifères [10]. La conservation d’une telle structure malgré le polymorphisme dans les séquences, suggère donc une pression de sélection importante et un rôle fonctionnel de cette architecture palindromique. La région 3’RR semble être l’élément régulateur apparu le plus récemment lors de l’évolution du locus IgH. Elle serait issue de la duplication de la région contenant les exons Cμ qui, ensuite, se serait elle-même dupliquée, formant ainsi la structure palindromique. Son apparition semble corrélée à celle de la recombinaison de classe chez les tétrapodes. Il est donc possible que son évolution, et les duplications successives, aient été facilitées par une pression de sélection imposant la mise en place d’une région régulatrice assurant le contrôle de ce mécanisme complexe [11]. Diverses publications ont rapporté le rôle de la région 3’RR dans son ensemble, mais aussi celui de chaque élément transcriptionnel, ainsi qu’une éventuelle synergie ou redondance entre ces éléments [1]. Schématiquement (
Figure 3
), la 3’RR n’est pas impliquée dans les phases précoces de la lymphopoïèse B (contraction du locus IgH, recombinaison DJH et VHDJH) [12, 13]. Sa structure palindromique est, par contre, essentielle au processus d’hypermutation somatique [14–16]. Ce sont ses activateurs transcriptionnels qui contrôlent son effet sur la recombinaison de classe [16–18] et la transcription des gènes constants du locus IgH [19]. La synergie entre les quatre éléments transcriptionnels est notable pour ces deux activités non seulement in vitro mais aussi in vivo. Enfin, au stade de B mature, la 3’RR prend l’activateur transcriptionnel Eµ sous sa domination transcriptionnelle, faisant de cette région, l’élément clef unique contrôlant tous les processus de transcription (sens, anti-sens, codant et non codant) au locus IgH [20]. |
L’origine des lymphoproliférations B Le développement et la prolifération des lymphocytes B sont des phénomènes étroitement contrôlés. Un déséquilibre de la balance entre apoptose et prolifération de ces cellules sera à l’origine de l’apparition de leucémies ou de lymphomes. Une cellule B ayant subi une transformation maligne conserve certains traits phénotypiques de sa cellule d’origine, ce qui permet d’identifier le précurseur à l’origine du lymphome et ainsi de le classer. Les lymphomes B représentent ainsi un groupe hétérogène comprenant plus de 40 sous-types, dont la majorité dérivent de cellules B matures [21]. La transformation d’une cellule fait intervenir plusieurs évènements oncogéniques qui modifient des fonctions en lien avec la prolifération cellulaire, l’apoptose ou encore l’interaction avec l’environnement. Dans le cas des lymphomes, il s’agit, d’une part, de lésions génétiques majoritairement aléatoires et, d’autre part, de translocations pouvant apparaître au gré des recombinaisons qui ponctuent l’ontogénie B [22, 23] (→). (→) Voir la Synthèse de S. Gisselbrecht, m/s n° 2, février 2003, page 201 Le développement des techniques de séquençage et de biologie moléculaire a permis de caractériser des translocations contribuant au développement de différentes tumeurs et s’avérant récurrentes. Plus récemment, les techniques de séquençage haut débit et de TC-seq (translocation capture-sequencing) ont permis une étude plus exhaustive des translocations et des mutations à l’origine de nombreux cancers, dont les lymphomes [22]. Ainsi, différents types de translocations sont retrouvés lors des lymphoproliférations B (
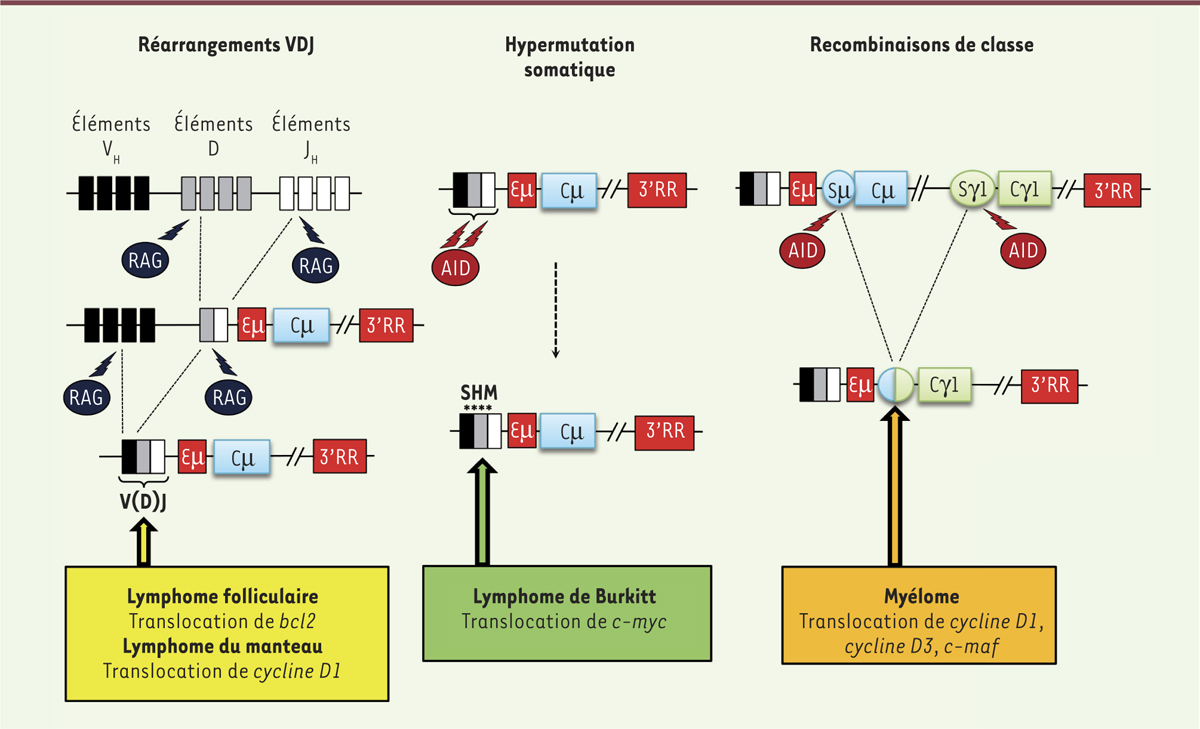
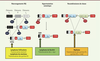
Figure 4
) et les plus fréquents conduisent à l’insertion au locus IgH de séquences codantes intactes de proto-oncogènes. Les exemples les plus connus sont la translocation du gène codant c-myc, associée au lymphome de Burkitt, de celui codant la cycline D1, dans le lymphome du manteau3, et celui codant Bcl2 (B-cell lymphoma 2), dans le lymphome folliculaire. Ces translocations sont généralement l’un des évènements les plus précoces de la transformation maligne. Elles ne sont toutefois pas suffisantes pour conduire à un cancer. Plusieurs évènements géniques (ou « hit ») seront nécessaires pour aboutir à une cancérisation complète de la cellule. Ces mutations qui permettent de contourner différents points de contrôle en affectant le cycle cellulaire, la résistance à l’apoptose, la signalisation intracellulaire, la reconnaissance par le système immunitaire, etc. [24], ne sont pas spécifiques des lymphoproliférations et sont retrouvées dans de nombreux types de cancers. C’est le cas, par exemple, de celles touchant le gène TP53, qui code la protéine suppresseur de tumeur p53, qui sont retrouvées dans près de la moitié des tumeurs chez l’homme. Ce facteur de transcription, parfois appelé « gardien du génome », contrôle l’arrêt du cycle cellulaire ou l’apoptose en réponse à un stress (lésion de l’ADN, défaut de réplication, etc.). Sa mutation est susceptible de créer un environnement permissif au développement de tumeurs.
 | Figure 4.
Translocations récurrentes au sein du locus IgH liées aux mécanismes de diversification des immunoglobulines. Les différents remaniements géniques qui ont lieu sur le locus IgH au cours du développement B en font un « point chaud » de translocation. Selon le mécanisme et les enzymes impliqués (RAG ou AID), la translocation peut avoir lieu dans les régions variables (flèche jaune et flèche verte) ou constantes (flèche orange) et impliquer différents proto-oncogènes. RAG : recombination-activating gene ; AID : activation-induced cytidine deaminase ; SHM : somatic hypermutation.
|
La majorité des lymphomes humains semblent issus de la transformation de cellules du centre germinatif (CG)4 ou post-CG. Cette observation peut sembler surprenante puisque les lymphocytes B ne résident que peu de temps dans cette zone du ganglion lymphatique, et que les lymphocytes B naïfs représentent plus de la moitié de l’ensemble des cellules B. Cela s’explique cependant par la prolifération intense des cellules du CG et par le déroulement de l’hypermutation somatique et de la recombinaison de classe, qui majorent considérablement le risque de mutations oncogéniques. La notion de cellule d’origine du lymphome doit cependant être nuancée. La cancérisation d’une cellule s’effectue en plusieurs étapes, pouvant affecter différents stades de développement de la cellule. Il est ainsi difficile de déterminer les stades de maturation auxquels les différentes altérations ont été acquises et l’impact qu’elles ont pu avoir sur le développement du lymphocyte B avant sa transformation maligne. Par exemple, dans les lymphomes folliculaires, le premier événement oncogénique consiste en une translocation plaçant le proto-oncogène BCL2 au sein du locus IgH. Cette translocation a généralement lieu lors du stade pro-B, mais elle n’entraînera le développement d’un lymphome que lors des stades tardifs du développement, durant lesquels l’expression de BCL2 est normalement réprimée [25]. C’est pour ces raisons que le terme « contrepartie normale » d’une cellule cancéreuse est souvent préféré à « cellule d’origine ». |
Les altérations oncogéniques au cours des lymphomes La plupart des lymphomes B se caractérisent par l’existence d’une translocation déplaçant un proto-oncogène au sein des locus d’immunoglobulines (Ig). Il sera alors transcrit de façon constitutive, sous le contrôle des éléments régulateurs propres à ces locus. La fréquence élevée de ces altérations géniques s’explique par l’induction de cassures doubles brins intervenant au cours des réarrangements qui ponctuent la lymphopoïèse et qui font des locus d’Ig des points chauds de translocations. Lors de certaines translocations, le point de cassure au sein du locus IgH peut être adjacent aux segments D ou J. La translocation résulte dans ce cas d’une erreur qui survient lors des réarrangements V(D)J. C’est le cas notamment des translocations BCL2-IgH ou CCND1 (le gène codant la cycline D1)-IgH [24]. Ces translocations nécessitent l’induction de deux cassures doubles brins (l’une dans le locus d’Ig, l’autre, à proximité du proto-oncogène). La cassure au locus IgH fait intervenir, dans la quasi-totalité des cas, l’enzyme RAG (recombination-activating gene) chargée d’induire les cassures doubles brins lors des recombinaisons V(D)J. La cassure à proximité du proto-oncogène peut, elle aussi, être induite par RAG (action hors cible), la translocation est alors due à un réarrangement VDJ illégitime. Cette cassure peut également être indépendante de RAG, l’extrémité du proto-oncogène allant alors envahir le complexe synaptique5 formé lors d’une recombinaison V(D)J légitime. Pour certaines translocations, les points de cassures se situent au sein, ou à proximité, de l’exon VDJ réarrangé. C’est, par exemple, le cas de la translocation c-MYC-IgH associée au lymphome de Burkitt endémique. Ce type de translocation implique l’enzyme AID et est caractéristique d’une erreur de réparation lors de l’hypermutation somatique. En effet, bien qu’elles ne soient pas nécessaires, des cassures doubles brins sont souvent induites au cours de l’hypermutation somatique, ce qui favorise l’apparition de translocations. Certaines translocations ciblent les régions S qui précèdent les exons codant les domaines constants. Elles proviennent de réparations illégitimes lors de la recombinaison de classe. C’est le cas par exemple des translocations c-MYC-IgH ou c-MAF-IgH, cette dernière étant associée aux myélomes multiples [22, 23, 25]. Une translocation requiert l’induction simultanée de deux cassures double brins. Si celles au locus IgH sont dues à l’action physiologique d’AID, celles à proximité du proto-oncogène peuvent provenir d’une activité hors cible de l’enzyme. En effet, malgré une régulation très stricte, AID peut agir sur certaines régions du génome de façon non spécifique. L’organisation spatiale nucléaire semble également jouer un rôle important, les translocations étant plus fréquentes entre des gènes localisés en cis ou des gènes situés à proximité, au sein du noyau [26, 27]. Dans le cas des lymphomes B, une proximité chromosomique importante a été décrite entre le gène CCND1 ou c-Myc et le locus IgH. Ce rapprochement des gènes varie au cours des différentes étapes de la maturation lymphocytaire B. Ainsi, dans les lymphocytes B naïfs, CCND1 est proche du locus IgH. Lors des étapes de maturation que sont l’hypermutation somatique et la recombinaison isotypique, c’est c-Myc qui est proche du locus IgH [28, 29]. La localisation nucléaire du chromosome transloqué est également modifiée. En effet, dans les cellules de lymphomes de Burkitt et du manteau, les locus c-Myc-IgH et CCND1-IgH sont relocalisés de la périphérie nucléaire (zone de faible transcription) vers la région centrale péri-nucléaire (zone de forte transcription) [30] où plusieurs protéines (dont LR1 [LPS-responsive factor] composant de la nucléoline) peuvent influencer l’activation des proto-oncogènes qui ont été transloqués [30, 31]. |
La 3’RR et la lymphomagénèse La translocation d’un proto-oncogène au locus IgH entraîne sa surexpression. Son contrôle transcriptionnel apparaît donc sous la dépendance des éléments régulateurs du locus IgH qui l’accueille. L’activateur intronique Eµ a été initialement soupçonné d’être l’acteur critique de cette induction d’expression des proto-oncogènes. Cependant, des souris transgéniques exprimant un transgène Eµ-c-Myc ne développent que des tumeurs de phénotypes immatures, différentes des lymphomes de Burkitt matures, retrouvés chez l’homme, ayant pour origine une translocation de c-Myc dans le locus IgH [32]. Ces résultats et le fait qu’Eµ puisse être éliminé lors des translocations c-Myc-IgH associées à des lymphomes de Burkitt sporadiques, ont conduit à suggérer que la région 3’RR puisse être responsable de la modulation d’expression du proto-oncogène transloqué. Cette implication de la 3’RR a en effet été validée dans des modèles murins : la transfection d’un transgène constitué d’une cassette c-Myc-3’RR [33], l’insertion d’une cassette 3’RR en 5’ du c-Myc endogène [34], ou l’insertion de c-Myc en divers points du locus IgH (en amont de Eµ ou de Cµ, ou dans Cα) [35–37], induisent l’apparition de lymphomes B similaires aux lymphomes de Burkitt humains et aux plasmocytomes. Le modèle de souris transfectées avec la cassette c-Myc-3’RR a révélé l’influence sur la lymphomagenèse, de mutations additionnelles (touchant TP53 ou CDK4 [cyclin-dependent kinase-4]) [38] et de l’isotype du BCR que les cellules expriment [39]. La confirmation définitive du rôle de la région 3’RR dans l’apparition des lymphomes a été apportée, chez la souris, par des expériences de délétions touchant les activateurs hs3b et hs4 qui favorisent leur développement : la 3’RR est en effet indispensable au développement de lymphomes matures mais pas de lymphomes pro-B [40]. Des études in vitro ont également prouvé que la 3’RR était responsable de la dérégulation de l’expression de BCL2 dans des lymphomes associés à une translocation BCL2-IgH [41]. Des expériences de capture de la conformation des chromosomes (3C)6 [46] (→) ont de plus permis de montrer la proximité spatiale (via des boucles d’activation de plusieurs méga-bases) entre les éléments activateurs de la 3’RR et le promoteur du BCL2 transloqué. (→) Voir la Synthèse de David Umlauf, m/s n° 3, mars 2015, page 304 L’ensemble de ces études a donc confirmé l’importance de la région 3’RR dans la lymphomagenèse B. Il montre également la pertinence des modèles murins pour l’étude de leur développement. Faisant suite à une translocation oncogénique au locus IgH, le « super-enhancer » 3’RR peut donc prendre sous sa dépendance transcriptionnelle, l’oncogène transloqué. Afin de remédier à la dérégulation de son expression ainsi générée, seul le ciblage pharmacologique de l’oncogène avait, jusqu’à présent, été envisagé [42, 43] (→). (→) Voir la Nouvelle de S. Pyndiah et D. Sakamuro, m/s n° 2, février 2013, page 133, et la Synthèse de Rémi Montagne et al., m/s n° 10, octobre 2014, page 864 Cibler spécifiquement dans la cellule B, l’activité transcriptionnelle de la 3’RR (qui repose sur ses activateurs), pourrait se révéler une stratégie thérapeutique prometteuse dans le cas des lymphomes B matures. L’activation de la 3’RR est sous le contrôle d’une histone déacétylase (HDAC) qui se lie spécifiquement sur l’activateur hs1,2. Les inhibiteurs d’HDAC (HDACi) régulent négativement l’activité de la 3’RR [32]. Ils apparaissent ainsi être une piste potentiellement intéressante en thérapeutique. Certaines de ces molécules capables, en théorie, de réprimer l’expression du locus IgH en agissant sur la 3’RR, sont actuellement en phase d’essai clinique pour le traitement de différents types de tumeurs (myélome, lymphomes du manteau, folliculaires ou diffus à grandes cellules). Elles présentent une cytotoxicité importante pour les cellules cancéreuses [44, 45]. Ces inhibiteurs sont-ils spécifiques de la région 3’RR ? Vraisemblablement pas ! Le seront-ils dans l’avenir ? Possiblement, mais pas dans un futur proche : les études sur l’inhibition des fonctions transcriptionnelles de la 3’RR n’en sont qu’à leurs balbutiements ! La réduction drastique du niveau d’expression des Ig en réponse aux HDACi via leur action sur la 3’RR laisse penser que ces molécules induiraient une baisse importante de la transcription du proto-oncogène transloqué au locus IgH. |
Les progrès à venir dans le traitement des lymphomes B reposeront, en grande partie, sur une meilleure connaissance de leur mode de développement. Ils pourront notamment résulter des études permettant d’élucider les mécanismes qui participent à la régulation du locus IgH, celui-ci paraissant être directement impliqué dans l’amplification de l’expression des proto-oncogènes transloqués. Les études récentes d’épigénétique et de transcriptomique ont révélé l’étendue du rôle de la région 3’RR dans les étapes clés de la maturation lymphocytaire B. Elle contrôle non seulement la transcription du locus IgH, mais aussi le remodelage chromatinien avec cassures multiples de l’ADN et l’élimination programmée de centaines de kilobases de matériel génétique. Si l’importance de cette fonction de la 3’RR dans la génération des lymphocytes B matures a été progressivement comprise au cours de ces dernières années, sa contrepartie délétère, lors de la lymphomagenèse B, apparaît de plus en plus évidente. Le rôle central de la 3’RR sur la transcription du locus IgH ne peut en effet qu’avoir des conséquences sur la transcription du proto-oncogène transloqué. Le rationnel de l’utilisation de drogues épigénétiques pour le traitement de certains lymphomes B matures n’était jusqu’ici que peu documenté. Leur effet négatif sur l’activation transcriptionnelle de la 3’RR dans le lymphocyte B pathologique avec, pour corolaire, la diminution de l’hypertranscription oncogénique, pourrait permettre d’expliquer les activités de ces drogues dans le traitement des lymphomes. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
Ce travail a été financé par l’ANR (projet EpiSwitch-3’RR 2016) et le Comité d’Organisation de la Recherche en Cancérologie du Limousin (CORC) (FJA/NP-2015-109). Nour Ghazzaui est financée par l’Association de spécialisation et d’orientation scientifique et la municipalité de Khiam (Liban).
|
Footnotes |
1.
Pinaud E, Marquet M, Fiancette R, et al. The IgH locus 3’ regulatory region: pulling the strings from behind . Adv Immunol. 2011; ; 110 : :27.–70. 2.
Perlot T, Alt FW, Bassing CH, et al. Elucidation of IgH intronic enhancer functions via germ-line deletion . Proc Natl Acad Sci USA. 2005; ; 42 : :14362.–14367. 3.
Marquet M, Garot A, Bender S, et al. The Eµ enhancer region influences H chain expression and B cell fate without impacting IgVH repertoire and immune response in vivo . J Immunol. 2014; ; 193 : :1171.–1183. 4.
Chauveau C, Cogné M. Palindromic structure of the IgH 3’ locus control region . Nat Genet. 1996; ; 14 : :15.–16. 5.
Birshtein BK. Epigenic regulation of individual modules of the immunoglobulin heavy chain 3’ regulatory region . Front Immunol. 2014; ; 5 : :163.. 6.
Garot A, Marquet M, Saintamand A, et al. Sequential activation and distinct functions for distal and proximal modules within the IgH 3’RR region . Proc Natl Acad Sci USA. 2016; ; 113 : :1618.–1623. 7.
Péron S, Laffleur B, Denis-Lagache N, et al. AID-driven deletion causes immunoglobulin heavy chain locus suicide recombination in B cells . Science. 2012; ; 336 : :931.–934. 8.
Péron S, Laffleur B, Denis-Lagache N, et al. Recombinaisons suicides du locus IgH : quand le lymphocyte B dépose les armes ! . Med Sci (Paris). 2012; ; 28 : :551.–554. 9.
Chen C, Birshtein BK. Virtually identical enhancers containing a segment of homology to murine 3’IgH-E(hs1,2) lie downstream of human Ig C alpha 1 and C alpha 2 genes . J Immunol. 1997; ; 159 : :1310.–1318. 10.
D’addabbo P, Scascitelli M, Giambra V, et al. Position and sequence conservation in Amniota of polymorphic enhancer HS1,2 within the palindrome of IgH 3’ regulatory region . BMC Evol Biol. 2011;; 11 : :71.. 11.
Magor BG, Ross DA, Pilström L, et al. Transcriptional enhancers and the evolution of the IgH locus . Immunol Today. 1999; ; 20 : :13.–17. 12.
Rouaud P, Vincent-Fabert C, Fiancette R, et al. Enhancers located in heavy chain regulatory region (hs3a, hs1,2, hs3b and hs4) are dispensable for diversity of VDJ recombination . J Biol Chem. 2012; ; 287 : :8356.–8360. 13.
Medvedovic J, Ebert A, Tagoh H, et al. Flexible long-range loops in the VH gene region of the IgH locus that likely facilitate the generation of a diverse antibody repertoire . Immunity. 2013; ; 39 : :229.–244. 14.
Rouaud P, Vincent-Fabert C, Saintamand A, et al. The IgH 3’ regulatory region controls AID-induced somatic hypermutation in germinal centre B-cells in mice . J Exp Med. 2013; ; 210 : :1501.–1507. 15.
Saintamand A, Vincent-Fabert C, Garot A, et al. Deciphering the importance of the palindromic architecture of the immunoglobulin heavy chain 3’ regulatory region . Nat Commun. 2016; ; 7 : :10730.. 16.
Le Noir S, Boyer F, Lecardeur S, et al. Functional anatomy of the immunoglobulin heavy chain 3’ super-enhancer needs not only core enhancer elements but also their unique DNA context . Nucleic Acids Res. 2017; ; 45 : :5829.–5837. 17.
Vincent-Fabert C, Fiancette R, Pinaud E, et al. Genomic deletion of the whole IgH 3’ regulatory region (hs3a, hs1,2, hs3b, hs4) dramatically affects class switch recombination and Ig secretion to all isotypes . Blood. 2010; ; 116 : :1895.–1898. 18.
Saintamand A, Rouaud P, Saad F, et al. Elucidation of IgH 3’ region regulatory role during class switch recombination via germline deletion . Nat Commun. 2015; ; 6 : :7084.. 19.
Saintamand A, Rouaud P, Garot A, et al. The IgH 3’ regulatory region governs µ chain transcription in mature B lymphocytes and the B cell fate . Oncotarget. 2015; ; 6 : :4845.–4852. 20.
Saintamand A, Vincent-Fabert C, Marquet M, et al. Eµ and 3’RR IgH enhancers show hierarchic unilateral dependence in mature B-cells . Sci Rep. 2017; ; 7 : :442.. 21.
Pasqualucci L, Dalla-Favera R. The genetic landscape of diffuse large B-cell lymphoma . Semin Hematol. 2015; ; 52 : :67.–76. 22.
Robbiani DF, Nussenzweig MC. Chromosome translocation, B cell lymphoma, and activation-induced cytidine deaminase . Ann Rev Pathol. 2013; ; 8 : :79.–103. 23.
Gisselbrecht S. Oncogènes et leucémies : historique et perspectives . Med Sci (Paris). 2003; ; 19 : :201.–210. 24.
Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation . Cell. 2011; ; 144 : :646.–674. 25.
Seifert M, Scholtysik R, Küppers R. Origin and pathogenesis of B cell lymphomas . Methods Mol Biol. 2013; ; 971 : :1.–25. 26.
Zhang Y, McCord RP, Ho YJ, et al. Spatial organization of the mouse genome and its role in recurrent chromosomal translocations . Cell. 2012; ; 148 : :908.–921. 27.
Nikiforova MN, Stringer JR, Blough R, et al. Proximity of chromosomal loci that participate in radiation-induced rearrangements in human cells . Science. 2000; ; 290 : :138.–141. 28.
Roix JJ, McQueen PG, Munson PJ, et al. Spatial proximity of translocation-prone gene loci in human lymphomas . Nat Genet. 2003; ; 34 : :287.–291. 29.
Sklyar I, Iarovaia OV, Gavrilov AA, et al. Distinct patterns of colocalization of the CCND1 and CMYC genes with their potential translocation partner IGH at successive stages of B-cell differentiation . J Cell Biochem. 2016; ; 117 : :1506.–1510. 30.
Allinne J, Pichugin A, Iarovaia O, et al. Perinucleolar relocalization and nucleolin as crucial events in the transcriptional activation of key genes in mantle cell lymphoma . Blood. 2014; ; 123 : :2044.–2053. 31.
Brys A, Maizels N. LR1 regulates c-myc transcription in B cell lymphomas . Pro Natl Acad Sci USA. 1994; ; 91 : :4915.–4919. 32.
Ghazzaui N, Saintamand A, Issaoui A, et al. The IgH 3’ regulatory region and c-myc induced B-cell lymphomagenesis . Oncotarget. 2017; ; 8 : :7059.–7067. 33.
Truffinet V, Pinaud E, Cogné N, et al. The 3’ IgH locus control region is sufficient to deregulate a c-myc transgene and promote mature B cell malignancies with a predominant Burkitt-like phenotype . J Immunol. 2007; ; 179 : :6033.–6042. 34.
Wang J, Boxer LM. Regulatory elements in the immunoglobulin heavy chain gene 3’-enhancers induce c-myc deregulation and lymphomagenesis in murine B cells . J Biol Chem. 2005; ; 280 : :12766.–12773. 35.
Park SS, Kim JS, Tessarollo L, et al. Insertion of c-Myc into IgH induces B-cell and plasma-cell neoplasms in mice . Cancer Res. 2005; ; 65 : :1306.–1315. 36.
Rosean TR, Holman CJ, Tompkins VS, et al. KSHV-encoded vIL6 collaborates with deregulated c-MYC to drive plasmablastic neoplasm in mice . Blood Cancer J. 2016; ; 6 : :e398.. 37.
Cheung WC, Kim JS, Linden M, et al. Novel targeted deregulation of c-myc cooperates with Bcl-XL to cause plasma cell neoplasms in mice . J Clin Invest. 2004; ; 113 : :1763.–1773. 38.
Rouaud P, Fiancette R, Vincent-Fabert C, et al. Mantle cell lymphoma-like lymphomas in c-myc-3’RR/p53+/- mice and c-myc-3’RR/Cdk4R24C mice: differential oncogenic mechanisms but similar cellular origin . Oncotarget. 2012; ; 3 : :586.–593. 39.
Amin R, Marfak A, Pangault C, et al. The class-specific BCR tonic signal modulates lymphomagenesis in a c-myc deregulation transgenic model . Oncotarget. 2014; ; 15 : :8995.–9006. 40.
Gostissa M, Yan CT, Bianco JM, et al. Long-range oncogenic activation of IgH-c-myc translocations by the IgH 3’ regulatory region . Nature. 2009; ; 462 : :803.–807. 41.
Duan H, Heckman CA, Boxer LM. The immunoglobulin heavy-chain gene γ’ enhancers deregulate bcl-2 promoter usage in t(14;18) lymphoma cells . Oncogene. 2007; ; 26 : :2635.–2641. 42.
Pyndiah S, Sakamuro D. Un trio de choc pour pallier la chimiorésistance des cancers : c-MYC, PARP1, BIN1 . Med Sci (Paris). 2013; ; 29 : :133.–135. 43.
Montagne R, Furlan A, Kherrouche Z, et al. Le récepteur Met fête ses 30 ans . Med Sci (Paris). 2014; ; 30 : :864.–873. 44.
Richardson PG, Mitsiades CS, Laubach JP, et al. Preclinical data and early clinical experience supporting the use of histone deacetylase inhibitors in multiple myeloma . Leuk Res. 2013; ; 37 : :829.–837. 45.
Morschhauser F, Terriou L, Coiffier B, et al. Phase 1 study of the oral histone deacetylase inhibitor abexinostat in patients with Hodgkin lymphoma, non-Hodgkin lymphoma, or chronic lymphocytic leukaemia . Invest New Drugs. 2015; ; 33 : :423.–431. 46.
Le Umlauf D. génome intime… et en trois dimensions . Med Sci (Paris). 2015; ; 31 : :304.–311. |