Les techniques de séquençage haut débit ont révolutionné l’identification des gènes impliqués dans la survenue de maladies génétiques [1] (→).
(→) Voir la Nouvelle de D. Stoppa-Lyonnet et C. Houdayer, m/s n° 2, février 2012, page 123
L’approche la plus utilisée est le séquençage d’exome, c’est-à-dire des exons, les séquences codantes du génome [2]. L’exome ne représente que 3 % du génome humain, mais son étude est particulièrement appropriée dans les maladies mendéliennes [3] (→).
(→) Voir la Nouvelle de A. Criqui et S. Baulande, m/s n° 5, mai 2010, page 452
La très grande majorité des mutations touche, en effet, les régions codantes, que ce soit des mutations faux-sens, non-sens ou des modifications d’épissage conduisant à un transcrit anormal ou absent. La puissance de cette technique est telle que des centaines de gènes ont pu être identifiés par séquençage de l’exome d’un seul patient, ou mieux du trio père-mère-enfant atteint d’une pathologie. Dans la moitié des cas environ, le séquençage d’exome ne permet cependant pas d’identifier la mutation responsable de la maladie pour différentes raisons : (1) certains exons sont mal couverts car ils sont riches en GC (guanine/cytosine) et difficiles à amplifier ; (2) les mutations modifient l’épissage, mais sont localisées loin dans l’intron ; ou (3) parce qu’il est souvent impossible d’identifier la mutation parmi tous les variants de signification inconnue (VUS : variants of unknown significance), mutation qui pourrait jouer sur la nature ou l’abondance de l’ARN messager (ARNm).
Récemment, le groupe d’Holger Prokisch, à Munich, a montré l’intérêt du séquençage du transcriptome (séquençage de l’ARN, ou RNA-seq) dans l’identification de gènes impliqués dans des maladies mendéliennes [4]. Le RNA-seq permet, en effet, de compléter l’information génétique brute donnée par le séquençage d’exome, par une donnée fonctionnelle qui révèle les conséquences de VUS, ou met en évidence des transcrits quantitativement ou qualitativement anormaux du fait d’une mutation dans des régions non-codantes. Cette étude a été menée sur une série de patients suspectés, sur des critères cliniques, d’avoir une maladie mitochondriale et pour lesquels le séquençage d’exome avait été infructueux.
Les maladies mitochondriales représentent collectivement les maladies métaboliques les plus fréquentes, mais leur extrême hétérogénéité clinique et génétique rend leur diagnostic génétique de plus en plus compliqué. Ces maladies résultent, en effet, de mutations touchant soit l’ADN mitochondrial (ou ADNmt), soit des gènes nucléaires responsables d’un très grand nombre de fonctions mitochondriales [5]. On connaît actuellement plus de 250 gènes nucléaires impliqués dans des maladies mitochondriales et des centaines de mutations de l’ADNmt. Les corrélations entre génotype et phénotype se révèlent parfois difficiles puisqu’un très petit nombre de patients partagent la même mutation génique. Enfin, le diagnostic d’une maladie mitochondriale repose le plus souvent sur la mise en évidence d’un déficit de la chaîne respiratoire mitochondriale (établi par l’analyse d’un tissu atteint : muscle, foie, cœur). Ce déficit enzymatique n’est pas toujours détectable dans les tissus accessibles et, parfois, il ne peut être vérifié, faute de matériel en quantité suffisante. Ces déficiences ne sont pas toujours retrouvées en culture, dans les fibroblastes provenant des biopsies. Leur interprétation nécessite également d’être mise en regard de l’expression et de l’évolution clinique de la maladie. L’extrême hétérogénéité de ces maladies et le chevauchement des signes cliniques avec ceux d’autres pathologies ont pu conduire à établir un faux diagnostic de maladie mitochondriale, constaté après l’identification du gène incriminé. Seule l’identification du gène muté permet ainsi d’affirmer le diagnostic.
Le groupe d’H. Prokisch a réalisé une analyse par RNA-seq à partir de fibroblastes provenant de 105 patients suspectés de développer une maladie mitochondriale, un séquençage d’exome avait auparavant été réalisé pour une partie d’entre eux. Les mitochondries sont présentes dans toutes les cellules, à l’exception des hématies, et les gènes codant des protéines mitochondriales ont une expression ubiquitaire, faisant des fibroblastes un bon matériel cellulaire pour ce type d’analyse et pour des études de protéomique. L’étude s’est attachée à identifier des gènes présentant un niveau d’expression anormal, un transcrit aberrant ou une expression monoallélique.
La puissance de cette approche est telle que très peu de gènes ont montré un niveau anormal d’expression dans chaque échantillon. Seuls trois échantillons ont présenté chacun un gène dont l’expression était indiscutablement diminuée, dont MGST1 (microsomal glutathione S-transferase 1), codant une glutathione S-transférase, pour un patient, et TIMMDC1 (translocase of inner mitochondrial membrane domain containing 1), pour deux autres patients, alors qu’aucune mutation de ces gènes n’avait été détectée par séquençage de l’exome. Dans tous les cas, l’étude protéomique a montré une diminution massive ou une absence de la protéine correspondante. TIMMDC1 code un facteur d’assemblage du complexe I de la chaîne respiratoire, et des études supplémentaires ont montré une diminution de l’expression des différentes sous-unités de ce complexe dans les fibroblastes de ces patients.
Un deuxième type de résultat concerne les gènes présentant des anomalies d’épissage [6] (→).
(→) Voir la Nouvelle de P. de la Grange, m/s n° 12, décembre 2016, page 1111
Dans ce cas également, peu de gènes comprenant des exons supplémentaires ou absents ont été détectés (175 en tout), et parmi les 20 montrant une anomalie d’épissage significative, deux codaient des protéines mitochondriales. L’analyse par RNA-seq a montré que le transcrit de TIMMDC1 comportait un exon supplémentaire à l’origine d’un décalage du cadre de lecture qui introduit un codon stop prématuré. Ce nouvel exon contenait un variant homozygote très rare, situé à 2 kilobases après l’exon 4, qui révèle un site cryptique d’épissage. Le transcrit contenant cet exon supplémentaire devenait alors instable. De façon plus classique, l’analyse par RNA-seq a également révélé la perte de l’exon 5 du gène CLPP (caseinolytic mitochondrial matrix peptidase proteolytic subunit) due à une variation homozygote de la dernière base de l’exon 5 qui avait été repérée par séquençage de l’exome mais classée comme VUS. L’absence de protéine CLPP normale dans les fibroblastes du patient a ensuite confirmé l’effet délétère de cette perte de l’exon 5.
Le dernier type d’anomalies implique des gènes à expression monoallélique. La comparaison des données du séquençage d’exome et du RNA-seq a permis de mettre en évidence 20 gènes définis comme contenant un ou des variants à l’état hétérozygote par le séquençage d’exome mais identifiés homozygotes par le RNA-seq, ce qui suggère l’absence d’expression d’un allèle. Parmi les 18 variants à expression monoallélique d’un patient, un seul VUS (une variation faux-sens) était localisé dans un gène codant une protéine mitochondriale, l’ALDH18A1 (aldehyde dehydrogenase 18, family member A1), impliquée dans le métabolisme de la proline. Le séquençage d’exome avait également mis en évidence une mutation non-sens hétérozygote dans le gène ALDH18A1, mais qui ne suffisait pas, à elle seule, à expliquer la maladie. Par la suite, une réduction majeure de la protéine ALDH18A1 a été observée. Le variant à expression monoallélique modifie la stabilité de la protéine et le gène ALDH18A1 est bien responsable de la maladie chez ce malade qui ne présentait pas tous les signes associés aux mutations du gène.
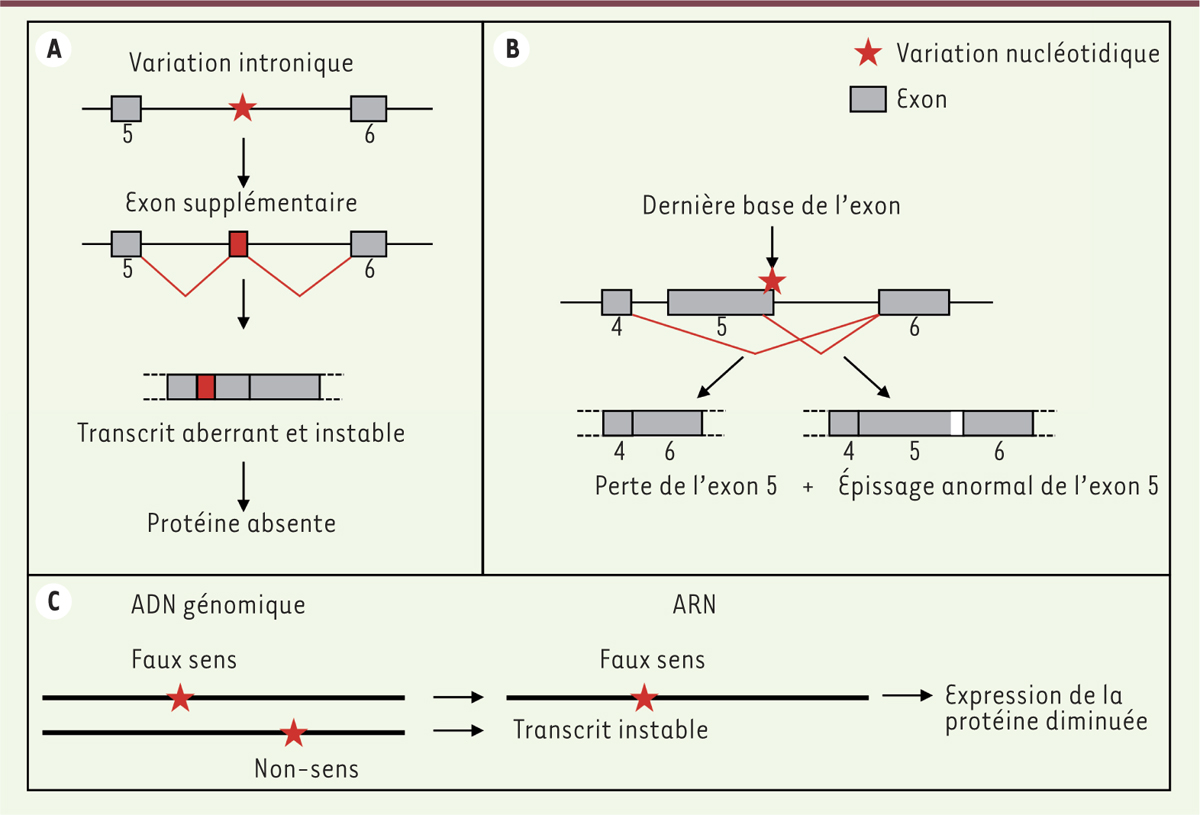
Ces différents exemples n’ont rien de révolutionnaire car on connaît depuis longtemps des exemples de variations nucléotidiques qui révèlent des sites cryptiques d’épissage, qui abolissent des sites d’épissage aux jonctions intron-exon, ou des mutations non-sens, qui conduisent à un transcrit instable (Figure 1), mais il fallait identifier le gène incriminé. La force du RNA-seq, en complément du séquençage d’exome, est de mettre en évidence ces mutations sans hypothèse préalable.
| Figure 1.
Exemples de variations nucléotidiques conduisant à des modifications quantitatives ou qualitatives des ARN messagers. A. Variation intronique révélant un site cryptique d’épissage et l’apparition d’un exon supplémentaire. B. Modification de la dernière base d’un exon induisant l’abolition d’un site d’épissage et la perte de l’exon. C. Expression monoallélique due à une variation conduisant à une mutation non-sens. |
Le RNA-seq s’avère donc être une approche relativement efficace qui permet de révéler des mutations indétectables par séquençage de l’exome, ou de démontrer l’effet de variations jusque-là sans signification connue. Comme toutes les approches à haut débit, le RNA-seq n’est pas suffisant, et le séquençage de l’exome, réalisé en parallèle, reste une première étape qui permet de résoudre environ la moitié des cas. Le développement d’outils bio-informatiques est fondamental pour l’interprétation des données massives qui sont générées par ces analyses. Les approches fonctionnelles seront également toujours nécessaires afin de valider les résultats obtenus et limiter les erreurs d’interprétation qui peuvent avoir des conséquences catastrophiques sur le conseil génétique et le diagnostic prénatal. Plusieurs des gènes identifiés dans les études que nous rapportons, avaient été identifiés auparavant chez d’autres patients présentant des phénotypes différents. Une nouvelle expertise clinique, a posteriori, devrait donc être envisagée dans ces cas. D’autres gènes identifiés codent en fait des protéines non-mitochondriales. Le diagnostic de maladie mitochondriale fondé sur l’étude enzymologique de la chaîne respiratoire nécessitera donc d’être évalué de nouveau. L’expertise clinique permettra de confirmer la maladie mitochondriale ou, au contraire, d’infirmer ce diagnostic. L’évolution et l’efficacité croissante des techniques de séquençage permettent d’accroître le nombre de diagnostics génétiques, mais ces techniques doivent être associées à une excellente expertise clinique qui aiguille et valide de façon rigoureuse l’interprétation des résultats.