| |
| Med Sci (Paris). 33(12): 1051–1054. doi: 10.1051/medsci/20173312011.La télomérase de la levure subit une cure de rajeunissement Nancy Laterreur1 and Raymund J. Wellinger1a 1Département de microbiologie et infectiologie, Faculté de médecine et des sciences de la santé, Université de Sherbrooke, 3201 Rue Jean Mignault Sherbrooke, Québec, J1E 4K8, Canada |
Un portrait nécessitant une cure de rajeunissement En 2009, le prix Nobel de physiologie ou médecine a été décerné à trois chercheurs : E.H. Blackburn, C.W. Greider et J.W. Szostak, pour leurs travaux sur les extrémités des chromosomes, les télomères, et sur une enzyme bien particulière : la télomérase. La télomérase est une enzyme essentielle au bon maintien et à la stabilité de notre génome et, aujourd’hui, nous savons qu’elle est composée de plusieurs protéines qui sont associées à un ARN, une organisation généralement conservée chez tous les organismes eucaryotes. L’importance biologique de la télomérase est confirmée également pour la survie des cellules cancéreuses, et pour son implication dans la sénescence des cellules humaines. Mais c’est pour les ramifications cliniques potentiellement très importantes de la télomérase qu’il existe un intérêt extraordinaire pour comprendre son mode d’action et sa régulation. La télomérase est connue depuis très longtemps. Cependant, toutes ses composantes ne sont probablement pas encore découvertes [1] (→).
(→) Voir la Nouvelle de F. Gallardo et P. Chartrand, m/s n° 3, mars 2009, page 232
Des cellules dérivées de plusieurs espèces ont été utilisées pour décrire le rôle de la télomérase, en particulier la levure bourgeonnante, Saccharomyces cerevisiae. Les nobélisés de 2009 l’ont tous utilisée, à un moment donné de leurs études. C’est d’ailleurs dans cet organisme que, récemment, de nouvelles protéines entrant dans la composition du complexe constituant la télomérase, ont pu être identifiées [2]. |
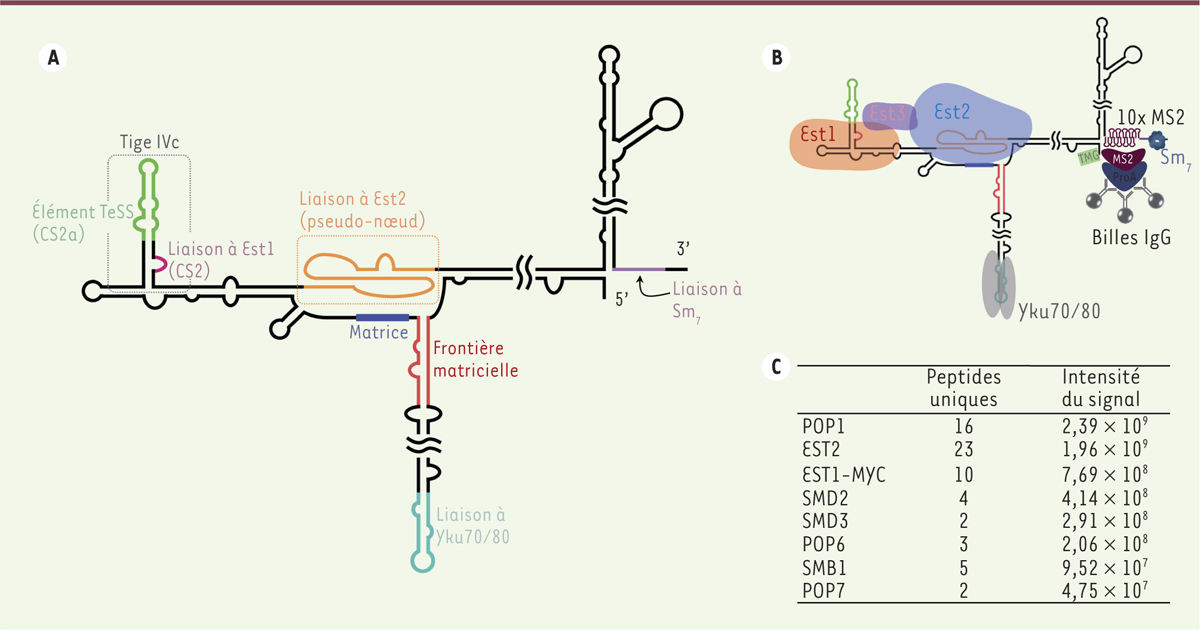
La télomérase : des protéines, oui, mais également un ARN ! Les ribonucléoprotéines (RNP) sont des complexes protéiques intégrant au moins une molécule d’ARN. La télomérase possède cette caractéristique : plusieurs protéines sont en effet regroupées sur une molécule d’ARN. Chez la levure, cet ARN se nomme Tlc1. C’est un long ARN non-codant d’environ 1 200 nucléotides. Trois protéines (Est1 [ever shorter telomere 1], Est2 et Est3) se lient à l’ARN Tlc1 et forment le cœur de l’enzyme. Est2 est la sous-unité qui génère l’activité catalytique de rétro-transcriptase [2]. Les protéines de la coiffe et de l’heptamère Sm7 se lient à l’ARN. Ils assurent sa stabilité et sa maturation. Finalement, l’hétérodimère yKu70/80 (une ADN hélicase dépendante de l’ATP) permet la localisation correcte du complexe [3]. L’ARN Tlc1 possède une structure secondaire très élaborée (Figure 1A) [4–6]. Plusieurs tiges-boucles et un pseudo-nœud contribuent à rendre cet ARN utile à l’activité catalytique de la télomérase. En fait, cette structure est plutôt flexible et certaines régions de l’ARN peuvent être déplacées ou même supprimées [7]. D’autres sont hautement conservées, en séquence et en structure, comme, par exemple, la région CS2/CS2a (conserved sequence 2/conserved sequence 2a) de la tige IVc [8]. Cette région était connue pour être nécessaire à la liaison de la protéine Est1 à l’ARN, elle s’avère être essentielle pour le recrutement de la télomérase sur les télomères [9]. L’élément CS2a est également important dans le maintien des télomères par la télomérase, mais sa fonction précise reste inconnue [10].
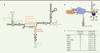 | Figure 1.
L’ARN Tlc1 est utilisé comme appât pour la purification de la télomérase, le complexe est ensuite analysé par spectrométrie de masse. A. Structure secondaire générale de Tlc1. On note, selon les couleurs, les régions importantes de l’ARN qui sont connues pour lier des protéines. Une exception : l’élément TeSS/CS2a (telomerase-stimulating structure/ conserved sequence 2a) de la tige IVc dont la contribution au complexe demeurait à déterminer. B. Schéma de la purification de la télomérase via l’ARN Tlc1. Dix tiges-boucles provenant de l’ARN génomique du bactériophage MS2 sont insérées à l’extrémité 3’ de l’ARN pour ainsi l’immunoprécipiter et entraîner les protéines qui y sont attachées. La protéine capside du phage MS2 (fusionnée à l’étiquette ProA) interagit avec les tiges-boucles MS2. Des billes couvertes d’immunoglobulines G (IgG) reconnaissent l’étiquette ProA et permettent l’immunopurification. C. Tableau présentant les 8 meilleurs candidats identifiés au cours de 3 expériences indépendantes de purification et d’analyse par spectrométrie de masse. La liste est organisée par ordre d’intensité du signal obtenu et le nombre de peptides retrouvés pour chacune d’entre elles est indiqué. |
Nos connaissances de l’organisation protéique de la télomérase, chez la levure, sont demeurées inchangées ces quinze dernières années. Cependant, de par sa très grande taille et sa structure, il était raisonnable de penser que l’ARN Tlc1 pouvait recruter d’autres protéines. La télomérase est réactivée dans plus de 90 % des cancers. La découverte de nouveaux acteurs interagissant avec l’ARN pourrait donc contribuer au développement de nouvelles cibles thérapeutiques. Considérant ces données, nous avons entrepris la purification de la télomérase de Saccharomyces cerevisiae, afin d’enrichir notre connaissance de ce complexe enzymatique. La purification de la télomérase a reposé sur le système MS2 [11] : dix tiges-boucles provenant de l’ARN génomique du bactériophage MS2 ont été insérées à l’extrémité 3’ de l’ARN Tlc1. La protéine de capside du phage MS2, qui reconnaît spécifiquement ces tiges-boucles, fusionnée avec la protéine A de la bactérie Staphylococcus aureus, appelée ProA, a été exprimée. Il est ainsi possible de purifier le complexe entier grâce à cette étiquette ProA, qui lie spécifiquement les immunoglobulines (Figure 1B). Après immobilisation sur des billes couplées aux immunoglobulines, l’extrait protéique est préparé pour la spectrométrie de masse (MS). En parallèle, un extrait de protéines dans lequel Tlc1 ne possède pas les tiges-boucles de MS2, a également été traité comme contrôle négatif, afin d’identifier les protéines qui seraient liées non-spécifiquement. |
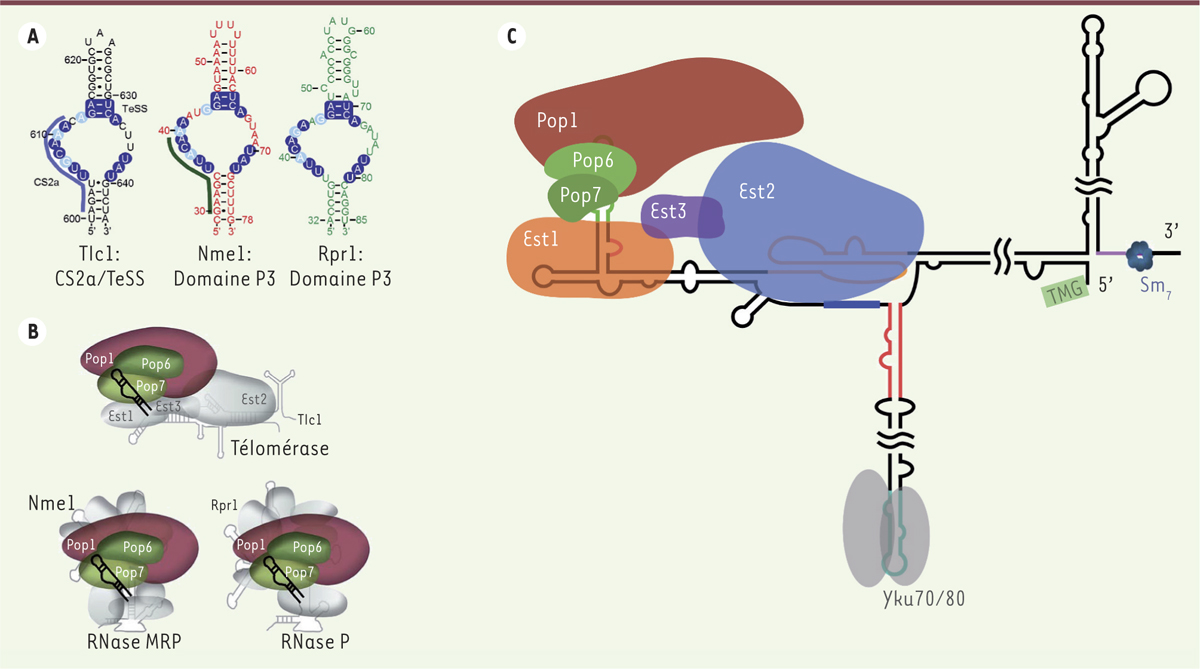
Peu de candidats, mais une agréable coïncidence Les résultats initiaux de MS ont identifié peu de candidats (Figure 1C). Parmi les plus abondants, se trouvaient des protéines connues pour lier l’ARN de la télomérase, comme Est1, Est2 et certaines protéines du complexe Sm7. Nous étions donc très confiants sur ces résultats, d’autant que ces protéines étaient identifiées de façon spécifique. L’analyse a également révélé d’autres candidats, inconnus comme partenaires de la télomérase : les ribonucléases Pop1 (processing of precursor 1), Pop6 et Pop7 (Figure 1C). Les protéines Pop1, Pop6 et Pop7 sont des sous-unités des complexes ribonucléases (RNase) P/MRP, chez la levure. Ces deux complexes essentiels sont hautement conservés et sont responsables du traitement des ARN de transfert (ARNt), des ARN ribosomaux (ARNr) et de certains ARN messagers (ARNm) [12]. Les deux complexes ribonucléases, P/MRP, contiennent, tout comme la télomérase, plusieurs sous-unités protéiques qui sont assemblées autour d’un ARN non-codant (nommés respectivement Nme1 pour la RNase MRP et Rpr1 pour la RNase P). L’analyse de la liaison de Pop1, Pop6 et Pop7 aux ARN permet de remarquer que Pop6 et Pop7 forment un hétérodimère qui lie une tige-boucle particulière, présente dans les deux ARN (Nme1 et Rpr1) et nommée domaine P3. La protéine Pop1, elle, est recrutée, ensuite, pour stabiliser le complexe entier. Elle entraînera la liaison des autres protéines partenaires des ribonucléases P/MRP [13, 14]. Une analyse précise de la structure de l’ARN Tlc1 montre que la région distale de la tige IVc, qui contient la séquence CS2a et le domaine TeSS (telomerase-stimulating structure), présente de fortes homologies avec le domaine P3 de Nme1 et Rpr1 (Figure 2A). En fait, les structures générales sont identiques et la séquence de la boucle principale est hautement conservée. Pour Nme1 et Rpr1, le contact avec l’hétérodimère Pop6/Pop7 est assuré par la portion N-terminale (nucléotides 30 à 40) de la boucle du domaine P3 [12]. Pour Tlc1, cette même région est essentielle à l’activité de la télomérase, sans que soit toutefois précisé s’il s’agit d’un site de liaison à des protéines [10].
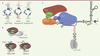 | Figure 2.
Le domaine P3 de Tlc1 possède les mêmes fonctions que celui des ARN Nme1 et Rpr1 des ribonucléases MRP/P : il lie l’hétérodimère Pop6/Pop7 pour ensuite recruter la protéine Pop1. A. Représentation de la portion distale de la tige IVc : le domaine TeSS (telomerase-stimulating structure) et la séquence CS2a (conserved sequence 2a) possèdent de fortes ressemblances avec les domaines P3 des ARN Nme1 et Rpr1. Les nucléotides (nt) identiques sont encerclés en bleu foncé. Les purines et pyrimidines conservées sont encerclées en bleu pâle. Le trait bleu à gauche de Tlc1 représente la portion essentielle à l’activité de la télomérase, l’élément CS2a. Le trait vert foncé à gauche de Nme1 représente les nucléotides protégés par la liaison de Pop6/Pop7. B. Les ARN de la télomérase (Tlc1), la ribonucléase (RNase) MRP (Nme1) et la RNase P (Rpr1) ont un élément commun : le domaine P3. Celui-ci est parfaitement interchangeable entre les trois ARN. C. Nouveau modèle de la télomérase chez la levure Saccharomyces cerevisiae. Le domaine P3 de Tlc1 recrute l’hétérodimère Pop6/Pop7. La protéine Pop1 se lie, par la suite, pour stabiliser le complexe de la télomérase formé des protéines Est1 (ever shorter telomere 1), Est2 et Est3. |
|
Tlc1 possède un domaine P3 liant les protéines Pop6/Pop7 Afin d’examiner si la portion distale de la tige IVc de Tlc1 présente les mêmes fonctionnalités que le domaine P3 de Nme1 et Rpr1, nous avons échangé les sous-domaines entre les différents ARN. La région de Tlc1 correspondant au domaine P3 a ainsi été insérée à la place du domaine P3 de Nme1, et les domaines P3 de Nme1 ou de Rpr1 ont remplacé le possible domaine P3 de Tlc1. Dans le premier cas, la RNase MRP (qui comporte l’ARN Nme1), essentielle chez la levure, une non-fonctionnalité de la portion dérivée de Tlc1 devrait se traduire par la mort des cellules. Les résultats obtenus montrent, en fait, une complète interchangeabilité entre le domaine P3 de Tlc1 et celui de Nme1. Inversement, les domaines P3 de Nme1 et de Rpr1 assurent une fonctionnalité totale de la télomérase lorsqu’ils remplacent la structure distale de la tige IVc de Tlc1, qui est essentielle à l’activité de l’enzyme télomérase (Figure 2B). Afin de démontrer que le domaine P3 de Tlc1 lie les protéines Pop6/Pop7 comme c’est le cas pour les RNases, des expériences de co-immunoprécipitation ont été réalisées. Dans des souches de levure exprimant une étiquette de reconnaissance sur une protéine du complexe Sm7 (Sme1) ainsi que sur les protéines Pop6 ou Pop7, nous avons pu démontrer qu’en l’absence du domaine TeSS/P3, l’ARN Tlc1 peut être co-immunoprécipité avec Sme1, mais qu’il n’entraîne pas les protéines Pop6 ou Pop7. Par ailleurs, l’ajout de la protéine Pop1, sous forme recombinante, à une réaction contenant Tlc1, Est2p et Pop6/Pop7, dans des expériences de reconstitution in vitro, augmente significativement l’activité de la télomérase. La protéine Pop1 serait donc nécessaire pour stabiliser le complexe enzymatique après la liaison de Pop6/Pop7. De nouvelles protéines entrent dans la constitution du complexe de la télomérase chez Saccharomyces cerevisiae (Figure 2C). Il est donc nécessaire d’actualiser l’ancien modèle et de le redessiner totalement. Ces découvertes soulèvent aussi plusieurs questions importantes. Est-ce un hasard si des complexes RNP considérés comme très anciens en termes d’évolution, partagent un tel mode de fonctionnement ? Pourquoi la télomérase nécessite-t-elle les protéines Pop6/Pop7 et Pop1 pour son fonctionnement alors que la télomérase et les ribonucléases jouent des rôles aussi différents au sein de la cellule? Quelle est la séquence des événements lors de l’assemblage du complexe ? Et, de façon plus large, l’association des protéines Pop à la télomérase est-elle conservée jusqu’aux vertébrés, et même les mammifères ? Cette découverte soulève beaucoup d’enthousiasme : l’identification de nouvelles protéines dans un complexe enzymatique aussi important que la télomérase pourrait permettre de mieux comprendre son mode d’action et, éventuellement de découvrir de nouvelles cibles thérapeutiques contre le cancer ! |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1.
Gallardo F, Chantrand P. Biogenèse de la télomérase : un long voyage jusqu’au bout des chromosomes . Med Sci (Paris). 2009; ; 25 : :232.–234. 2.
Lemieux B, Laterreur N, Perederina A, et al. Active yeast telomerase shares subunits with ribonucleoproteins RNase P and RNase MRP . Cell. 2016; ; 165 : :1171.–1181. 3.
Wellinger RJ, Zakian VA. Everything you ever wanted to know about Saccharomyces cerevisiae telomeres: beginning to end . Genetics. 2012; ; 191 : :1073.–1105. 4.
Forstemann K, Lingner J. Telomerase limits the extent of base pairing between template RNA and telomeric DNA . EMBO Rep. 2005; ; 6 : :361.–366. 5.
Zappulla DC, Cech TR. Yeast telomerase RNA: A flexible scaffold for protein subunits . Proc Natl Acad Sci USA. 2004; ; 101 : :10024.–10029. 6.
Dandjinou AT, Levesque N, Larose S, et al. A phylogenetically based secondary structure for the yeast telomerase RNA . Curr Biol. 2004; ; 14 : :1148.–1158. 7.
Zappulla DC, Cech TR. RNA as a flexible scaffold for proteins: yeast telomerase and beyond . Cold Spring Harb Symp Quant Biol. 2006; ; 71 : :217.–224. 8.
Gunisova S, Elboher E, Nosek J, et al. Identification and comparative analysis of telomerase RNAs from Candida species reveal conservation of functional elements . RNA. 2009; ; 15 : :546.–559. 9.
Seto AG, Livengood AJ, Tzfati Y, et al. A bulged stem tethers Est1p to telomerase RNA in budding yeast . Genes Dev. 2002; ; 16 : :2800.–2812. 10.
Laterreur N, Eschbach SH, Lafontaine DA, Wellinger RJ. A new telomerase RNA element that is critical for telomere elongation . Nucleic Acids Res. 2013; ; 41 : :7713.–7724. 11.
Gallardo F, Laterreur N, Cusanelli E, et al. Live cell imaging of telomerase RNA dynamics reveals cell cycle-dependent clustering of telomerase at elongating telomeres . Mol Cell. 2011; ; 44 : :819.–827. 12.
Esakova O, Krasilnikov AS. Of proteins and RNA: the RNase P/MRP family . RNA. 2010; ; 16 : :1725.–1747. 13.
Perederina A, Esakova O, Quan C, et al. Eukaryotic ribonucleases P/MRP: the crystal structure of the P3 domain . EMBO J. 2010; ; 29 : :761.–769. 14.
Perederina A, Esakova O, Koc H, et al. Specific binding of a Pop6/Pop7 heterodimer to the P3 stem of the yeast RNase MRP and RNase P RNAs . RNA. 2007; ; 13 : :1648.–1655. |