 |
| |||
Med Sci (Paris). 33(12): 1055–1062. doi: 10.1051/medsci/20173312012.La modélisation mathématique, un outil essentiel pour l’étude du ciblage thérapeutique des tumeurs solides 1Laboratoire d’oncobiologie moléculaire, Centre de biologie humaine (CBH), CHU Amiens Sud, Amiens, France 2Laboratoire amiénois de mathématique fondamentale et appliquée (LAMFA), CNRS UMR7352, UFR des sciences, Université de Picardie Jules Verne, Amiens, France 3Laboratoire de biochimie, Centre de biologie humaine (CBH), CHU Amiens Sud, Amiens, France 4Équipe CHIMERE (Chirurgie et extrémité céphalique, caractérisation morphologique et fonctionnelle), Université de Picardie Jules Verne, Amiens, France Corresponding author. | ||||
| ||||
Le cancer est une maladie complexe, caractérisée par l’occurrence d’anomalies allant du niveau moléculaire au corps entier. L’accumulation des mutations et les altérations touchant la régulation de l’expression de gènes contribuent à la transformation maligne [1]. L’issue de la maladie résulte des perturbations des systèmes qui régissent la transduction du signal dans la cellule cancéreuse, de la compétition entre cellules malignes et cellules saines, et de la capacité de la tumeur à coopter l’angiogenèse, à envahir les tissus et à former des métastases, et à échapper aux mécanismes effecteurs de l’immunité antitumorale [2]. Ces dernières décennies, les avancées de la génomique ont permis d’identifier des acteurs moléculaires essentiels de la cancérogenèse dont certains, comme les composants de la voie oncogénique RTK (receptor tyrosine kinase) – Ras – Raf – MEK (mitogen-activated ERK kinase) – ERK (extracellular signal-regulated kinase), constituent des cibles thérapeutiques potentielles [3]. La conception d’inhibiteurs spécifiques de ces protéines a permis de faire la preuve de principe de l’utilité de ces recherches avec, par exemple, le ciblage thérapeutique de B-Raf1 dans le mélanome malin [4]. Malheureusement, malgré un rationnel biologique établi, ces traitements anticancéreux présentent une efficacité qui se révèle le plus souvent temporaire. Après une période de temps limitée, des adaptations de la machinerie cellulaire peuvent apparaître et, dans certains cas, se traduire par l’inversion de l’effet du médicament, le transformant en un stimulant de la progression tumorale [5]. Des travaux récents suggèrent que ces remarquables propriétés d’adaptation peuvent découler de l’organisation intrinsèque des réseaux biologiques [6]. Il est donc nécessaire de prendre en compte l’organisation en réseaux et la régulation dynamique des cibles biologiques : c’est un des intérêts du recours à la modélisation mathématique [7–9] (→). (→) Voir la Synthèse de Emmanuel Barillot et al., m/s n° 6-7, juin-juillet 2009, page 601 Nous présentons dans cette revue l’apport de la modélisation mathématique pour les études d’oncobiologie moléculaire et, plus précisément, pour l’étude de la transduction du signal dans la cellule cancéreuse. | ||||
Les étapes essentielles et les questions importantes pour la construction et l’utilisation des modèles mathématiques appliqués à l’étude du cancer, sont résumées dans la Figure 1. Schématiquement, les modèles mathématiques sont répartis selon la nature et le caractère prévisible des évènements qui sont modélisés, en modèles stochastiques ou en modèles déterministes [10]. Les modèles déterministes reposent sur l’utilisation d’équations différentielles ordinaires (EDO) et d’équations aux dérivées partielles (EDP). Les équations différentielles stochastiques (EDS) décrivent, elles, les évènements aléatoires. Les modèles mathématiques sont également différenciés en modèles continus et discrets. Les modèles continus sont adaptés à l’étude de la dynamique des phénomènes biologiques et donc, aux réponses des cellules cancéreuses dans le temps. Dans une optique formelle, les approches de modélisation sont souvent présentées selon deux catégories : (1) le « Bottom-up » : à partir d’éléments d’un système, une intégration des données est proposée afin de comprendre la fonctionnalité de l’ensemble. Ceci exige des connaissances préalables sur le système et nécessite que les interactions moléculaires et leurs paramètres cinétiques soient bien caractérisés. Les connaissances concernant les composants cellulaires sont ensuite fusionnées pour former un modèle plus large. Cette approche est particulièrement applicable à la biologie moléculaire et à la biochimie pour confirmer un comportement dans des cellules intactes, par exemple, l’organisation générale des grands réseaux de transduction oncogénique ; (2) le « Top-down », également connu sous le nom de reverse engineering. Cette approche convertit notamment les données issues du « big data » en connaissances sur un système. Elle est devenue plus courante avec la disponibilité accrue de données à grande échelle obtenues par les méthodes de métagénomique par exemple. Par la modélisation, le système est examiné afin de déterminer les principaux événements qui le régissent. Idéalement, la modélisation mathématique est conçue en parallèle d’expériences menées in vitro ou in vivo. Des allers-retours successifs entre ces deux approches (mathématique et biologique) permettent ainsi de construire et d’enrichir le modèle. Il sera validé lorsque les principales conclusions qui ont été émises in silico auront été confirmées expérimentalement. Il est important de mentionner que la réussite, comme l’échec, d’un modèle mathématique peuvent être informatifs. Il peut en effet être intéressant de révéler que les connaissances biologiques prises en compte pour la modélisation étaient insuffisantes. La modélisation n’est donc pas une fin en soi, mais une approche qui sera associée en parallèle, à des études de biologie [11] (→). (→) Voir la Nouvelle de B. Martin et F. Mahé, page 1035 de ce numéro
À l’heure actuelle, les équations différentielles ordinaires (EDO) représentent l’outil le plus largement utilisé pour les modélisations en biologie. Elles sont typiquement adaptées aux analyses quantitatives du comportement d’un nombre limité d’acteurs biologiques [12]. Une limitation importante à leur utilisation réside dans le fait qu’elles sont réservées à l’étude d’un petit nombre d’acteurs : les modèles reposant sur les EDO les plus larges incluent en effet une trentaine d’acteurs pour lesquels des données numériques sont disponibles [12]. D’autres formalismes mathématiques2 sont applicables lorsque les données ne sont pas adaptées aux EDO, par exemple lorsqu’il s’agit de données semi-quantitatives ou qualitatives. | ||||
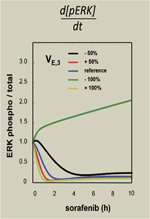
Les approches utilisées pour étudier les voies de signalisation intracellulaires reposent souvent sur l’application d’équations différentielles ordinaires (EDO) qui permettent d’en étudier la dynamique. Ces équations peuvent, par exemple, être construites à partir de la loi d’action de masse, ou de la cinétique de Michaelis-Menten, quand les interactions entre composants de ces voies sont de nature enzymatique [12]. La voie Raf-MEK-ERK est l’un des exemples de transduction oncogénique les plus étudiés sous le prisme mathématique, en raison de son implication fréquente dans le cancer [3, 9]. La première modélisation mathématique de cette voie oncogénique, centrée sur l’étude dynamique des composants Raf, MEK, et ERK, a été rapportée en 1996 [13]. Ce travail et plusieurs autres réalisés depuis, ont permis d’explorer des aspects importants de la régulation dynamique de cette cascade de transduction dans les cellules eucaryotes [12]. Parmi les contributions les plus notables, il a été possible d’expliquer (1) la cinétique d’activation hypersensible, en « tout ou rien », de ERK observée à l’échelle de la cellule unique [14], (2) d’explorer l’effet des protéines d’« échafaudage »3 [15], (3) d’expliquer les cinétiques distinctes d’activation de ERK selon les contextes de prolifération/différenciation cellulaire [16], ou encore (4) de mieux comprendre comment l’activation de ERK est convertie en régulation transcriptionnelle [17]. Nous avons étudié la voie Raf-MEK-ERK dans les cellules de carcinome hépatocellulaire (CHC) exposées au sorafénib4 [18] (Figure 2). Des EDO non-linéaires traduisant une cinétique enzymatique ont été utilisées pour modéliser les interactions, dans plusieurs lignées de CHC, entre les principaux acteurs de la voie Raf : B-Raf, C-Raf, MEK et ERK. Notre approche associant biologie et mathématiques a identifié deux points importants : (1) la grande hétérogénéité, selon les lignées de cellules, des modes de régulation de la signalisation en aval de Raf : l’analyse de sensibilité5, révèle, en effet, le rôle de régulateurs essentiels de la voie Raf, différents dans chacune des lignées ; (2) l’importance d’une étape insoupçonnée : l’étape de déphosphorylation des kinases ERK par les phosphatases. En l’absence de phosphatases spécifiques de MEK et de ERK, le sorafénib perd sa capacité de bloquer ERK dans des cellules pourtant sensibles à ce médicament [18]. Cette observation, quoique surprenante, le sorafénib ayant été conçu et développé comme inhibiteur de kinases, a pu être validée expérimentalement. Les approches de modélisation permettent donc de comprendre comment est régulée une voie oncogénique, comme la voie ERK, dans un contexte thérapeutique [18]. Ce type d’études permet secondairement, de guider la recherche de biomarqueurs originaux. Elles pourraient être utiles pour prédire l’efficacité du traitement et personnaliser la prescription du sorafénib. La modélisation mathématique révèle donc les détails de l’architecture en réseau des kinases Raf-MEK-ERK. Ces informations cruciales dans le contexte thérapeutique peuvent être obtenues par modélisation, même lorsque le réseau étudié est constitué d’un petit nombre d’acteurs (ici, les kinases Raf, MEK et ERK) [18, 19].
Pour les réseaux complexes, qui peuvent inclure des milliers d’acteurs, il est nécessaire d’appliquer des stratégies de réduction de la complexité, comme l’analyse modulaire (modular response analysis, ou MRA). La première étape de ce type d’analyse repose sur l’identification des nœuds, c’est-à-dire des composants du système qui sont fortement connectés. Cette identification peut être fondée sur des connaissances antérieures du système, ou découler d’études statistiques (par exemple, par regroupement hiérarchique). La dynamique entre les nœuds peut ensuite être analysée à l’aide d’EDO. Pour chaque paramètre, un coefficient de réponse locale, qui reflète l’impact d’un nœud sur l’autre, est défini [20, 21]. Ce type d’analyses a été appliqué, par exemple, à la modélisation des interactions entre les voies oncogéniques Raf-MEK-ERK et AKT (ou protéine kinase B)-mTOR (mammalian target of rapamycin), dans des cellules de cancer colorectal. Elles ont permis de proposer un ciblage thérapeutique amélioré, en associant l’inhibition de MEK et de l’EGFR (epidermal growth factor receptor)6, [20]. Ce type d’approche permet donc d’étudier l’impact des multiples boucles de rétrocontrôle au sein de larges réseaux de transduction. | ||||
Les exemples de modélisation des niveaux d’activation des kinases oncogéniques que nous avons présentés reposent sur des analyses de populations cellulaires. Ils font donc abstraction du comportement individuel des cellules. À l’échelle de la cellule, les voies oncogéniques, comme ERK, sont activées préférentiellement sous la forme de pics [22, 23], apparaissant de façon partiellement stochastique, avec une fréquence qui conditionne les réponses des cellules, comme la mitogenèse [22, 23]. L’état d’activation des composants d’amont (le « bruit », qui est une caractéristique des réseaux biologiques), mais aussi les différentes concentrations des acteurs de la cascade de signalisation, rendent impossible une modélisation mathématique entièrement déterministe. La modélisation de cette composante stochastique permet d’explorer les différences de régulation dynamique de Raf-MEK-ERK entre cellules, et d’en trouver l’origine. On peut ainsi montrer l’importance des différences qui existent entre cellules, de génomes identiques, et qui s’exercent en amont du module MEK-ERK [24, 25]. Cette notion d’hétérogénéité cellulaire intra-tumorale est importante. Elle conditionne en effet de nombreux aspects de la progression tumorale et la réponse des tumeurs aux traitements médicaux [26]. Les travaux de modélisation des voies oncogéniques à l’échelle des cellules uniques révèlent l’existence d’une hétérogénéité phénotypique d’origine non-génétique, inhérente à l’organisation des réseaux de transduction au sein des cellules cancéreuses [24, 25]. Les modèles qui incorporent une composante stochastique permettent donc de décrire, et de comprendre en partie, cette hétérogénéité phénotypique qui caractérise les tumeurs. | ||||
Des approches utilisant la logique booléenne, permettent également d’étudier les réseaux de transduction dans les cellules cancéreuses [27–29]. Sur le principe, l’analyse quantitative est, dans ce type de modèle, remplacée par des variables binaires qui décrivent la fonctionnalité de chaque élément : l’activation de chaque acteur d’un réseau est soumise à différentes conditions logiques, et la stabilité des réseaux booléens dépend des connections qu’établissent les nœuds qui les constituent. Ce type de modèle présente l’avantage de permettre l’étude de nombreux acteurs, généralement choisis à partir de connaissances préexistantes dans le domaine. L’un des objectifs de ce type de modélisation est d’identifier les états attracteurs, présumés représenter les états stables du système modélisé. Ces états stables peuvent, par exemple, correspondre aux différents statuts du réseau oncogénique dans un contexte de ciblage thérapeutique. L’étude de la régulation de la protéine p537,, un des déterminants de l’efficacité des chimiothérapies anti-tumorales, offre un bon exemple d’application de la modélisation logique en oncologie [27] (Figure 3). Les modèles booléens peuvent également permettre d’appréhender certains mécanismes à l’origine de la plasticité phénotypique observée dans les cellules cancéreuses. En définissant les états attracteurs dans un système booléen, il est possible d’aborder l’existence de plusieurs états métastables8, au sein d’un système biologique [29]. Une étude menée sur les cancers du poumon à petites cellules, offre un exemple récent d’utilisation d’un modèle booléen fondé sur l’état d’activation de 33 facteurs de transcription. Il a en effet été possible dans ce cas de mettre en évidence plusieurs états du réseau transcriptionnel et de les relier à l’émergence d’une hétérogénéité phénotypique dans le contexte thérapeutique [30].
Des approches pratiques permettent de contourner une limitation essentielle des modèles booléens qui tendent à refléter grossièrement la réalité biologique. Une étude récente rapporte ainsi l’intérêt d’un formalisme à l’interface entre logique booléenne et EDO afin de modéliser la dynamique des réseaux [31]. Cette étude, qui révèle l’existence de différences individuelles entre des lignées cellulaires cancéreuses coliques, montre également que l’information contenue dans l’organisation en réseau prédit la sensibilité des cellules à un panel de médicaments de ciblage thérapeutique, indépendamment de l’analyse génomique [31]. Ce type de modélisation apporte donc une information utile à la compréhension des mécanismes qui rendent les cellules cancéreuses sensibles ou résistantes au ciblage thérapeutique. | ||||
Les études que nous relatons apportent la preuve de principe que la modélisation mathématique permet d’étudier le fonctionnement dynamique des voies de transduction oncogéniques, et que cette information est utile afin d’identifier des modalités de ciblage thérapeutique qui soient optimisées. Désormais, un objectif essentiel de la modélisation mathématique consiste en la conception d’outils qui permettront d’apprécier, pour chaque patient, cette organisation dynamique et de reconstruire in silico le réseau de transduction oncogénique qui lui est propre, ceci dans l’optique du développement d’une médecine personnalisée et prédictive [32]. Il est donc important, après l’étape préclinique, d’envisager désormais des études clinico-biologiques. Ceci suppose d’appliquer la modélisation établie dans les lignées de cellules, aux réseaux de transduction du signal présents dans les tumeurs des patients, à partir des données biologiques accessibles dans le contexte clinique. Ces données sont souvent limitées, notamment parce qu’elles nécessitent la réalisation de biopsies. Les mathématiques offrent des solutions pratiques à cette limitation. Une étude récente montre comment il est possible de tirer parti des stratégies mathématiques d’optimisation combinatoire9 pour exploiter les données de phospho-protéomique obtenues à partir de biopsies de glioblastomes [33] : associée aux connaissances préexistantes des réseaux d’interaction entre protéines, et avec un nombre limité de marqueurs analysés, la modélisation a permis de reconstruire in silico les voies de transduction activées dans ces tumeurs [33]. Un deuxième objectif essentiel consistera à valider le caractère prédictif des modèles mathématiques, en les comparant notamment aux analyses « classiques », centrées sur les éléments isolés constituant les réseaux modélisés [34]. À long terme, modéliser en clinique les voies de transduction oncogénique devrait contribuer à favoriser l’émergence de l’oncologie prédictive [32]. L’utilisation des modèles mathématiques permet l’intégration de données obtenues à différents niveaux. L’oncologie prédictive repose ainsi sur la conception de larges modèles multi-échelle, qui combinent la modélisation mathématique des voies oncogéniques, et celle décrivant les interactions entre clones cellulaires [35–38], avec des aspects spécifiques, comme la pharmacocinétique/pharmacodynamique des médicaments [39]. Au final, la modélisation mathématique constitue une piste prometteuse pour optimiser les recherches biologiques et faciliter la transposition en clinique des connaissances qui en sont issues. | ||||
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. | ||||
Les auteurs remercient le comité de la Somme de la Ligue contre le Cancer, le CHU d’Amiens, et le Conseil régional des Hauts de France pour leur soutien à la recherche. | ||||
1
Une des trois protéines Raf. Les mutations touchant BRAF sont trouvées dans différents types de tumeurs.
2
Se dit d’un ensemble de règles et propriétés mathématiques qui sont utilisées dans un cadre précis.
3
Les protéines d’échafaudage sont considérées comme des réacteurs chimiques sur lesquels les molécules impliquées dans le transfert des signaux viennent se fixer simultanément avec l’orientation qui leur permet de se rencontrer et d’interagir.
4
Le sorafénib a une action bloquante sur les protéines C-Raf et B-Raf. Il est le médicament de référence pour le carcinome hépatocellulaire.
5
Permet d’identifier les paramètres et les variables d’entrée qui ont une forte influence sur les sorties d’un modèle.
6
La signalisation induite par l’EGFR implique PI3K (phosphatidyl inositol-1,4,5-trisphosphate 3-kinase) à l’origine de l’activation d’AKT et de mTOR.
7
La protéine p53 est l’un des plus importants suppresseurs de tumeur. Le gène TP53 qui la code est muté dans près de la moitié des cas de cancers humains.
| ||||
1.
Martincorena I, Campbell PJ. Somatic mutation in cancer and normal cells . Science. 2015; ; 349 : :1483.–1489. 2.
Egeblad M, Nakasone ES, Werb Z. Tumors as organs: complex tissues that interface with the entire organism . Dev Cell. 2010; ; 18 : :884.–901. 3.
Fey D, Matallanas D, Rauch J, et al. The complexities and versatility of the RAS-to-ERK signalling system in normal and cancer cells . Semin Cell Dev Biol. 2016; ; 58 : :96.–107. 4.
Chapman PB, Hauschild A, Robert C, et al. Improved survival with vemurafenib in melanoma with BRAF V600E mutation . N Engl J Med. 2011; ; 364 : :2507.–2516. 5.
Das Thakur M, Salangsang F, Landman AS, et al. Modelling vemurafenib resistance in melanoma reveals a strategy to forestall drug resistance . Nature. 2013;; 494 : :251.–255. 6.
Jordan NV, Bardia A, Wittner BS, et al. HER2 expression identifies dynamic functional states within circulating breast cancer cells . Nature. 2016; ; 537 : :102.–106. 7.
Barillot E, Calzone L, Zinovyev A. Biologie des systèmes appliquée aux cancers . Med Sci (Paris). 2009; ; 25 : :601.–607. 8.
Janes KA, Lauffenburger DA. Models of signalling networks: what cell biologists can gain from them and give to them . J Cell Sci. 2013; ; 126 : :1913.–1921. 9.
Kolch W, Halasz M, Granovskaya M, Kholodenko BN. The dynamic control of signal transduction networks in cancer cells . Nat Rev Cancer. 2015; ; 15 : :515.–527. 10.
Altrock PM, Liu LL, Michor F. The mathematics of cancer: integrating quantitative models . Nat Rev Cancer. 2015; ; 15 : :730.–745. 11.
Martin B, Mahé F. Un modèle mathématique pour l’étude des interactions bactériennes en biofilm Med Sci (Paris). 2017; ; 33 : :1035.–1038. 12.
Orton RJ, Sturm OE, Vyshemirsky V, et al. Computational modelling of the receptor-tyrosine-kinase-activated MAPK pathway . Biochem J. 2005; ; 392 : :249.–261. 13.
Huang CY, Ferrell JE Jr. Ultrasensitivity in the mitogen-activated protein kinase cascade . Proc Natl Acad Sci USA. 1996; ; 93 : :10078.–10083. 14.
Ferrell JE Jr, Machleder EM. The biochemical basis of an all-or-none cell fate switch in Xenopus oocytes . Science. 1998; ; 280 : :895.–898. 15.
Levchenko A, Bruck J, Sternberg PW. Scaffold proteins may biphasically affect the levels of mitogen-activated protein kinase signaling and reduce its threshold properties . Proc Natl Acad Sci USA. 2000; ; 97 : :5818.–5823. 16.
Sasagawa S, Ozaki Y, Fujita K, Kuroda S. Prediction and validation of the distinct dynamics of transient and sustained ERK activation . Nat Cell Biol. 2005; ; 7 : :365.–373. 17.
Nakakuki T, Birtwistle MR, Saeki Y, et al. Ligand-specific c-Fos expression emerges from the spatiotemporal control of ErbB network dynamics . Cell. 2010; ; 141 : :884.–896. 18.
Saidak Z, Giacobbi AS, Louandre C, et al. Mathematical modelling unveils the essential role played by cellular phosphatases in the inhibition of RAF-MEK-ERK signalling by sorafenib in Hepatocellular carcinoma cells . Cancer Lett. 2017; ; 392 : :1.–8. 19.
Jensen KJ, Moyer CB, Janes KA. Network architecture predisposes an enzyme to either pharmacologic or genetic targeting . Cell Syst. 2016; ; 2 : :112.–121. 20.
Klinger B, Sieber A, Fritsche-Guenther R, et al. Network quantification of EGFR signaling unveils potential for targeted combination therapy . Mol Syst Biol. 2013; ; 9 : :673.. 21.
Halasz M, Kholodenko BN, Kolch W, Santra T. Integrating network reconstruction with mechanistic modeling to predict cancer therapies . Sci Signal. 2016;; 9 : :ra114.. 22.
Albeck JG, Mills GB, Brugge JS. Frequency-modulated pulses of ERK activity transmit quantitative proliferation signals . Mol Cell. 2013; ; 49 : :249.–261. 23.
Voliotis M, Perrett RM, McWilliams C, et al. Information transfer by leaky, heterogeneous, protein kinase signaling systems . Proc Natl Acad Sci USA. 2014; ; 111 : :E326.–E333. 24.
Ryu H, Chung M, Dobrzyn´ski M, et al. Frequency modulation of ERK activation dynamics rewires cell fate . Mol Syst Biol. 2015; ; 11 : :838.. 25.
Filippi S, Barnes CP, Kirk PD, et al. Robustness of MEK-ERK dynamics and origins of cell-to-cell variability in MAPK signaling . Cell Rep. 2016; ; 15 : :2524.–2535. 26.
Meacham CE, Morrison SJ. Tumour heterogeneity and cancer cell plasticity . Nature. 2013; ; 501 : :328.–337. 27.
Abou-Jaoudé W, Ouattara DA, Kaufman M. From structure to dynamics: frequency tuning in the p53-Mdm2 network I . Logical approach. J Theor Biol. 2009; ; 258 : :561.–577. 28.
von der Heyde S, Bender C, Henjes F, et al. Boolean ErbB network reconstructions and perturbation simulations reveal individual drug response in different breast cancer cell lines . BMC Syst Biol. 2014; ; 8 : :75.. 29.
Abou-Jaoudé W, Traynard P, Monteiro PT, et al. Logical modeling and dynamical analysis of cellular networks . Front Genet. 2016; ; 7 : :94.. 30.
Udyavar AR, Wooten DJ, Hoeksema M, et al. Novel hybrid phenotype revealed in small cell lung cancer by a transcription factor network model that can explain tumor heterogeneity . Cancer Res. 2017; ; 77 : :1063.–1074. 31.
Eduati F, Doldàn-Martelli V, Klinger B, et al. Drug resistance mechanisms in colorectal cancer dissected with cell type-specific dynamic logic models . Cancer Res. 2017; ; 77 : :3364.–3375. 32.
Yankeelov TE, Quaranta V, Evans KJ, Rericha EC. Toward a science of tumor forecasting for clinical oncology . Cancer Res. 2015; ; 75 : :918.–923. 33.
Tuncbag N, Milani P, Pokorny JL, et al. Network modeling identifies patient-specific pathways in glioblastoma . Sci Rep. 2016; ; 6 : :28668.. 34.
Fey D, Halasz M, Dreidax D, et al. Signaling pathway models as biomarkers: Patient-specific simulations of JNK activity predict the survival of neuroblastoma patients . Sci Signal. 2015;; 8 : :ra130.. 35.
Marusyk A, Tabassum DP, Altrock PM, et al. Non-cell-autonomous driving of tumour growth supports sub-clonal heterogeneity . Nature. 2014; ; 514 : :54.–58. 36.
Almendro V, Cheng YK, Randles A, et al. Inference of tumor evolution during chemotherapy by computational modeling and in situ analysis of genetic and phenotypic cellular diversity . Cell Rep. 2014; ; 6 : :514.–527. 37.
Chisholm RH, Lorenzi T, Lorz A, et al. Emergence of drug tolerance in cancer cell populations: an evolutionary outcome of selection, nongenetic instability, and stress-induced adaptation . Cancer Res. 2015; ; 75 : :930.–939. | ||||