| |
| Med Sci (Paris). 34(2): 137–144. doi: 10.1051/medsci/20183402011.La biogenèse des ARN courts non codants chez les animaux Lucile Fressigné1,2** and Martin J. Simard1* 1Groupe St-Patrick de recherche en oncologie fondamentale, Oncologie-Centre de recherche du CHU de Québec-université Laval (L’hôtel-Dieu de Québec) ; Centre de recherche sur le cancer, université Laval, 9 rue McMahon, G1R3S3 Québec, Canada 2Programme de biologie moléculaire et cellulaire de la faculté de médecine, université Laval, 9 rue McMahon, G1R3S3 Québec, Canada |
Vignette (Photo © Inserm - Pascal Heitzer). Ces dernières années, la découverte des ARN courts non codants a ouvert un tout nouveau champ de la biologie moléculaire. En effet, ces petites séquences, ne codant pour aucune protéine, agissent comme de puissants régulateurs de l’expression des gènes. Il existe différents types d’ARN courts non codants, les mieux caractérisés étant les microARN, les piARN (Piwi-interacting ARN) et les siARN (small interfering ARN). Du fait de leur importante fonction d’ajustement dans la régulation des gènes et dans l’expression du génome, une mauvaise régulation du niveau d’expression de ces courts ARN peut entraîner l’apparition de nombreuses pathologies. Cette revue se concentre sur la biogénèse de ces ARN courts non codants chez les animaux. |
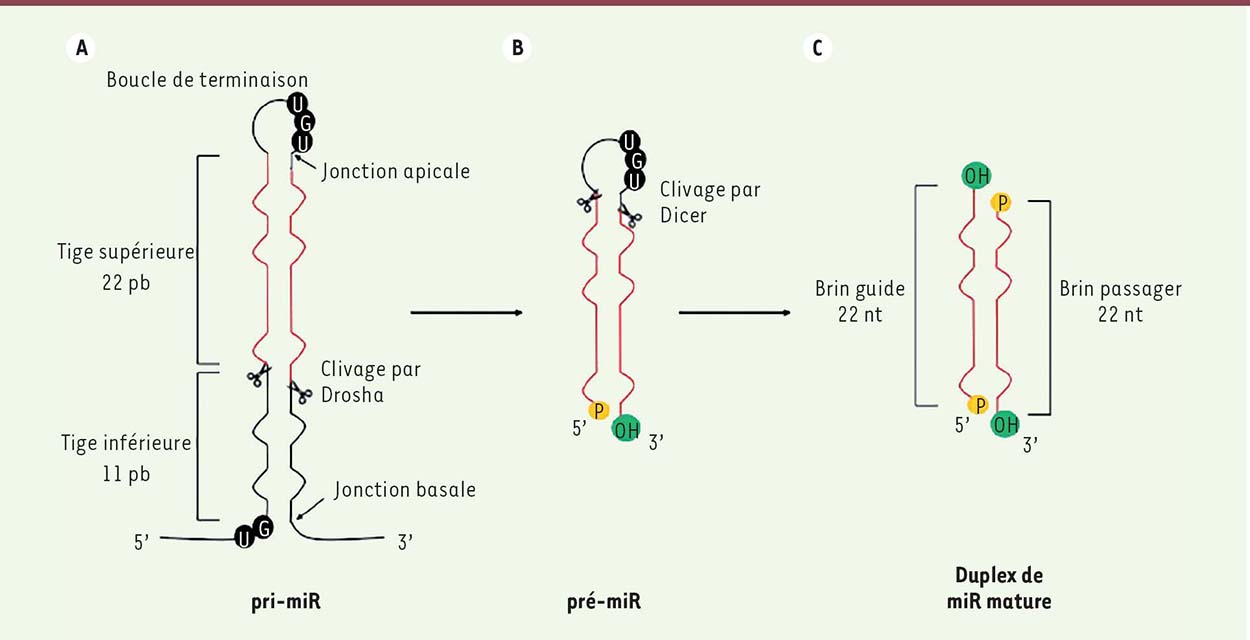
La voie de biogenèse des microARN Les microARN (miARN), d’une longueur de 21 à 23 nucléo-tides, représentent la famille d’ARN courts non codants la mieux caractérisée. C’est en 1993 que le premier miARN, lin-4, a été découvert chez le nématode Caenorhabditis elegans par le groupe de Victor Ambros [1]. Depuis, ils ont été décrits comme étant bien conservés au cours de l’évolution, des plantes jusqu’à l’homme, suggérant une fonction biologique importante. Les miARN régulent l’expression génique en se liant à la région 3’ non-traduite (3’ UTR ou 3’ untranslated region) des ARN messagers (ARNm) [2] (➔). (➔)Voir la Synthèse de Y. Romero et al., m/s n° 5, mai 2012, page 490 De nombreuses études montrent qu’ils sont impliqués dans divers processus biologiques comme le développement et la différenciation cellulaire. C’est pourquoi une mauvaise régulation de leur niveau d’expression peut entraîner différentes pathologies telles que le cancer. Organisation génomique des miARN Les miARN de différentes espèces sont répertoriés dans une base de données en ligne appelée miRBase 1, un outil puissant et complet permettant d’étudier la génomique des miARN. Chez l’homme, 54 % des miARN sont produits à partir de régions inter-géniques, les miARN restants sont codés dans les régions introniques des gènes codant une protéine [ 3]. Chez C. elegans et la drosophile, respectivement seulement 15 % et 39 % des miARN, sont produits à partir de régions introniques [ 4]. Les miARN peuvent être transcrits sous forme mono ou poly-cistronique. Les transcrits poly-cistroniques produisent plusieurs miARN à partir d’un seul transcrit formant ainsi un groupe de miARN. Chez C. elegans, la souris et l’homme, la majorité de ces ARN courts non codants sont retrouvés isolés et non en groupe. Le plus souvent, ces groupes codent des miARN faisant partie de la même famille, ce qui signifie qu’ils possèdent la même séquence clé de reconnaissance à l’ARNm, appelée « seed region ». Transcription des miARN La première étape dans la production des miARN est la transcription du miARN primaire (ou pri-miR) dans le noyau. Ces pri-miR, de plus de 1 kilobase, sont majoritairement transcrits par l’ARN polymérase II en une structure en forme de tige-boucle (ou épingle à cheveux) présentant des extrémités simple brin en 5’ et 3’ (Figure 1A). La jonction entre les extrémités simple brin et la tige centrale du pri-miR est appelée région basale. Des études à large échelle de génomique et de bioinformatique ont permis de localiser les sites d’initiation de la transcription, communément appelés TSS (transcription start site), des miARN chez les mammifères. L’une des premières études réalisées afin de définir les frontières génomiques des transcrits primaires de miARN, a permis de localiser les TSS jusqu’à une distance de 2 000 paires de bases en amont des miARN intergéniques [ 5]. Une étude de cartographie des nucléosomes a dévoilé les promoteurs de 175 miARN et a démontré que plus d’un tiers des miARN localisés dans les régions introniques, avaient un TSS différent de celui du gène hôte [ 6]. Il est maintenant établi que les sites d’initiation de la transcription peuvent se retrouver à une distance pouvant aller jusqu’à 40 kilobases en amont des régions de précurseurs de miARN [7-9]. Grâce à l’évolution des techniques, un atlas des miARN et de leur promoteur a été réalisé. Il fait partie intégrante du projet FANTOM5 ( functional annotation of mammalian genome) [ 3]. Maturation du transcrit primaire À la suite de sa transcription, s’ensuit la première étape de maturation constituée par le clivage du pri-miR en un précurseur de miARN (le pré-miR) d’environ 65 nucléotides. Ce clivage est réalisé dans le noyau par une enzyme de la famille des ARNases (ou ribonucléases) de type III, appelée Drosha. Cette dernière, possédant une activité endonucléase spécifique à l’ARN double brin, clive la structure tige boucle du pri-miR constituée, cette fois-ci, d’une boucle de terminaison et d’une tige double brin (Figure 1A). Mais pour effectuer un clivage précis, Drosha nécessite un cofacteur, DGCR8 (DiGeorge syndrome critical region 8) aussi appelé Pasha (partner of Drosha) chez la drosophile et PASH-1 chez C. elegans, les deux protéines formant un complexe appelé microprocesseur. Des études récentes ont montré que Drosha se lie au motif UG présent à la jonction basale tandis qu’un dimère de DGCR8 interagit avec la tige du pri-miR et reconnaît le motif UGU présent à la jonction apicale [ 10] (Figure 2). En plus de son activité, Drosha « mesure » la distance précise, à partir de la jonction basale, de 11 paires de bases où aura lieu le clivage, tandis que le dimère de DGCR8 stabilise l’enzyme [ 10]. Le pré-miR ainsi formé présente deux nucléotides flottants à son extrémité 3’ hydroxyle (OH) ainsi qu’un groupement phosphate à son extrémité 5’ (Figure 1B).Export cytoplasmique du pré-miR Après le clivage par le microprocesseur, le pré-miR est exporté vers le cytoplasme où il subira une deuxième étape de maturation (Figure 2). Chez les mammifères et la drosophile, l’exportine 5 (XPO5), un récepteur de transport nucléaire, associée à une protéine liant le GTP, Ran-GTP, forme un complexe qui, en se liant spécifiquement aux deux nucléotides flottants de l’extrémité 3’ du pré-miR, et en hydrolysant le GTP, va permettre la libération du pré-miR dans le cytoplasme [11, 54, 55]. Ce modèle de libération, fondé sur des essais de liaisons et des expériences de diminution de l’expression de XPO5, a été remis en question par de récents travaux. Il a en effet été montré qu’une perte d’expression de XPO5 n’affectait que modestement le niveau d’expression des miARN ce qui suggère que XPO5 n’est pas indispensable à la voie de biogenèse [ 12]. Chez C. elegans (qui ne possède par d’orthologue de XPO5), XPO-1 semble impliqué dans l’export des pré-miR, mais le mécanisme d’action exact reste à définir [ 56]. Maturation du pré-miR Dans le cytoplasme, les pré-miR sont pris en charge par Dicer (Dicer-1 chez la drosophile), une ARNase de type III, afin d’être clivés en duplex d’ARN de 22 nucléotides (Figures 1C et 2). Dicer reconnaît spécifiquement les deux nucléotides flottants en 3’-OH, l’extrémité 5’ phosphate, ainsi que la boucle de terminaison qu’elle excise (Figure 1). Dicer s’associe à des protéines liant l’ARN double brin comme PACT (protein activator of PKR) et TRBP (trans-activation response RNA-binding protein) chez l’homme, et Loquacious, chez la drosophile (D. melanogaster). Encore aujourd’hui, le rôle de ces protéines dans la voie des miARN n’est pas précisé. 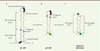 | Figure 1. Structure des formes primaire, précurseur et mature du miARN. A. Les pri-miR sont majoritairement transcrits par l’ARN polymérase II mais certains sont transcrits par l’ARN polymérase III. Ils sont constitués d’une boucle de terminaison, d’une tige et de deux extrémités simple brin. Drosha, aidé de DGCR8 (DiGeorge syndrome critical region 8), « mesure » 11 paires de bases (pb) à partir de la jonction basale et clive le pri-miR à cette position. B. Le pré-miR est constitué d’une boucle de terminaison, d’une tige, d’une extrémité 5’ présentant un groupement phosphate (P) et de deux nucléotides flottants à l’extrémité 3’ hydroxyle (3’OH). Dicer reconnaît les extrémités 5’ et 3’ par son domaine PAZ (un domaine de liaison aux acides nucléiques retrouvé chez les protéines Dicer et Argonautes). La boucle de terminaison est alors alignée le long de l’axe de Dicer qui « mesure » 22 nucléotides (nt) à partir des extrémités et clive la boucle de terminaison. C. Le duplexe de miARN mature compte 22 nt et est constitué du brin guide et du brin passager. |
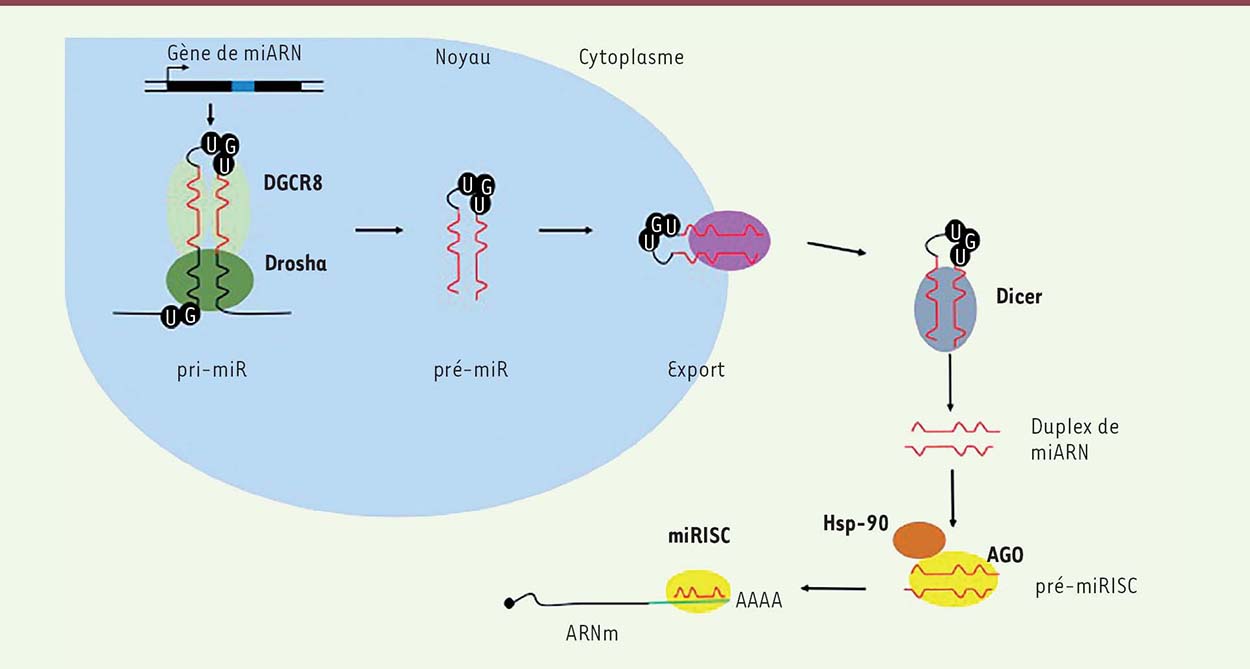
Un modèle alternatif de maturation a également été proposé. Dans ce modèle, le pré-miR, pris en charge par le complexe Dicer, TRBP et Ago2 (protéine Argonaute), est clivé à son extrémité 3’ par Ago2 en un ac-pré-miR (Ago2-cleaved precursor miRNA), lui-même clivé par Dicer [13]. Cet ac-pré-miR pourrait permettre une meilleure sélection du brin guide, brin du duplex d’ARN, qui est gardé pour former le miRISC (microRNA-induced silencing complex) [13]. Ce modèle a été décrit principalement pour le miARN let-7, et n’a pas été validé pour les autres miARN. Chez C. elegans, il a été montré que l’activité de clivage des protéines Argonautes ALG-1/2, est importante pour coordonner la production du miARN avec Dicer [14]. Formation du miRISC, le complexe effecteur RISC est un complexe ribonucléoprotéique effecteur, qui est appelé miRISC lorsqu’il comprend un miARN. L’assemblage du miRISC se réalise en deux étapes. La première consiste en la formation du complexe pré-miRISC (precursor miRISC), qui permet le transfert du produit de clivage de Dicer aux protéines Argonautes, des protéines majeures du complexe effecteur. Chez les mammifères, il existe quatre protéines Argonaute impliquées dans la voie des miARN (Ago1 à 4), alors que C. elegans en compte deux, ALG-1 et ALG-2, et D. melanogaster, une seule, Ago1 ([ 15]). Le pré-miRISC est constitué des protéines Dicer, Argonautes et les chaperonnes moléculaires Hsc (heat shock cognate protein) 70 / Hsp (heat shock protein) 90. Ces dernières, en hydrolysant l’ATP, permettent d’« ouvrir » la protéine Argonaute afin de faciliter la prise en charge du duplex d’ARN [ 16]. L’importance de l’action des chaperonnes dans la dynamique de l’assemblage du pré-miRISC a été montrée chez la drosophile [ 17], mais il reste a définir si ce mécanisme est conservé chez les autres espèces animales. La deuxième étape consiste en la séparation des brins d’ARN afin de ne garder que le brin guide au sein du mi RISC et d’éliminer le brin passager, qui sera dégradé. La sélection du brin guide est fondée sur la stabilité thermodynamique des extrémités 5’ du duplex : le brin présentant l’extrémité 5’ la moins stable sera conservé [18, 19]. En revanche, le mécanisme de séparation des deux brins reste inconnu. Après l’élimination du brin passager, le complexe minimal du miRISC, constitué d’une protéine Argonaute et du brin guide, est alors formé, prêt a assurer sa fonction de répression (Figure 2). | Figure 2. Voie de biogenèse des miARN. Le pri-miR est transcrit par l’ARN polymérase II. Il est ensuite clivé par le microprocesseur constitué de Drosha/DGCR8 (DiGeorge syndrome critical region 8) pour former le pré-miR. Ce dernier est exporté dans le cytoplasme pour être de nouveau clivé par Dicer. Le duplexe de miARN mature ainsi formé est alors pris en charge par une protéine Argonaute (AGO) aidée de la protéine chaperonne Hsp-90 (heat shock protein 90). Le brin passager est dégradé, le brin guide restant ancré dans la protéine Argonaute pour former le miRISC. Celui-ci pourra s’associer à ces ARNm cibles afin de réprimer leur traduction. |
Production de miARN non canoniques Des voies alternatives de biogenèse des miARN, dites non-canoniques, indépendantes de Drosha ou Dicer ont été décrites. La première a avoir été caractérisée est celles des mirtrons, des miARN fonctionnels dont la maturation contournent l’étape réalisée par Drosha, et qui sont produits par la machinerie d’épissage [ 20]. Ces miARN épissés adoptent une structure en tige-boucle semblable a celle des pré-miR. Ils peuvent, par la suite, intégrer la voie canonique des miARN [ 20]. Un miARN, le miR-451, est produit indépendamment de Dicer. Ce miARN d’abord clivé par Drosha (comme décrit précédemment), est exporté dans le cytoplasme où il est directement pris en charge par Ago2 qui va le cliver pour former le duplex de miARN mature [21, 22]. |
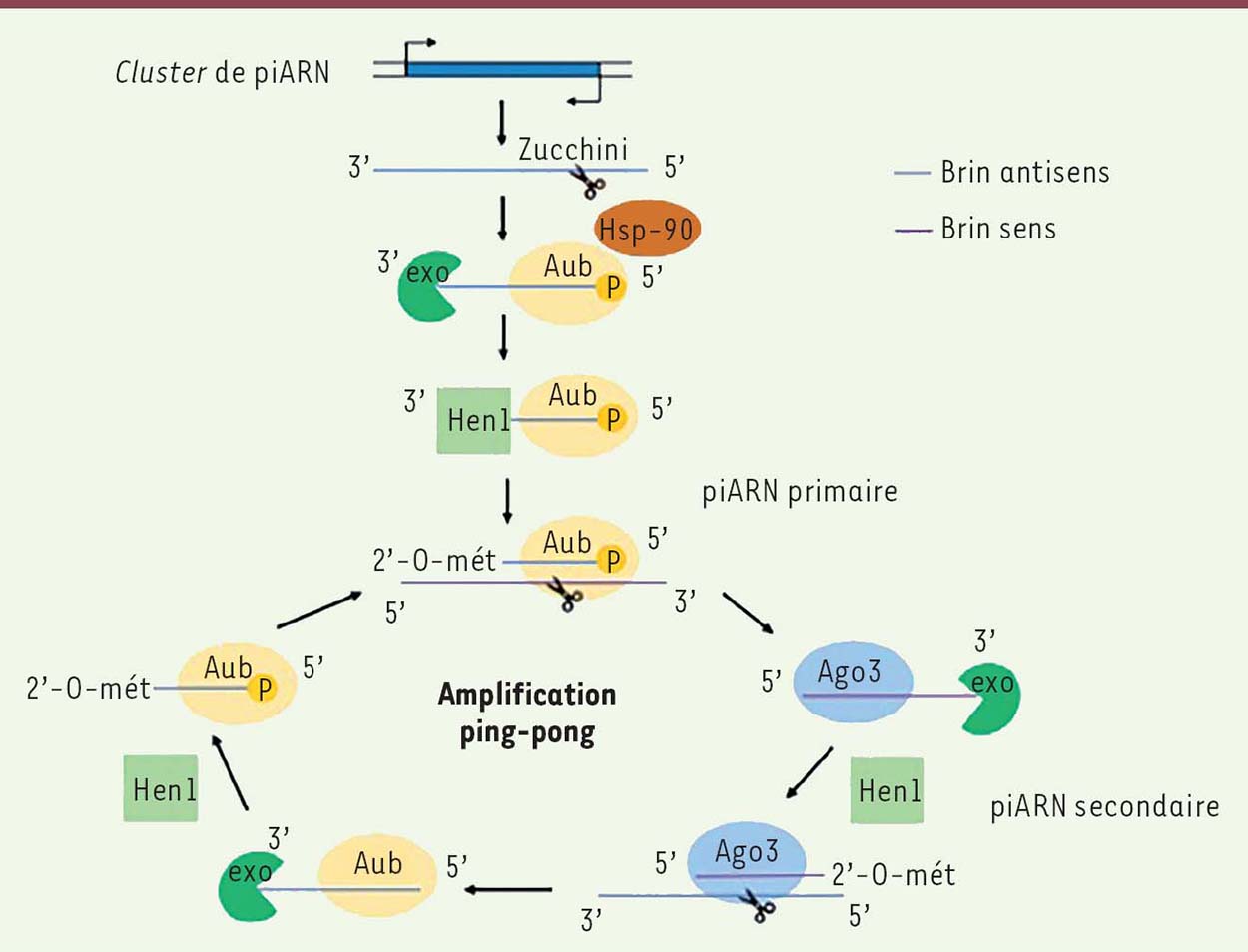
Les PIWI-interacting RNA ou piARN Les piARN représentent la famille des ARN courts non codants la moins caractérisée. Ces mystérieux ARN courts non codants présentent une méthylation sur l’oxygène en 2’ de leur extrémité 3’, ainsi qu’une base uridine à leur extrémité 5’. Ils sont exprimés spécifiquement dans la lignée germinale des animaux. Leur rôle est de protéger les cellules germinales d’« envahisseurs » du génome, comme les éléments transposables [23], en s’associant à des protéines Argonautes spécialisées, appelées PIWI. Plusieurs travaux ont montré l’existence d’une faible quantité de piARN également dans les cellules somatiques. Leur rôle dans la régulation des processus somatiques reste cependant à découvrir d’autant que, dans ces cellules, leur association aux protéines PIWI est encore inconnue [24]. Chez la souris, trois PIWI sont impliquées dans la voie des piARN, MILI (PIWIL2), MIWI (PIWIL1) et MIWI2 (PIWIL4), alors que chez C. elegans, il en existe deux, PRG-1/2, et trois chez la drosophile, PIWI, Aubergine (Aub) et AGO3 ([15]). La fonction des piARN est conservée au cours de l’évolution. Ils sont cependant utilisés différemment selon les espèces afin de réprimer les transposons. Biogenèse des piARN chez la drosophile et la souris La voie de biogenèse des piARN chez la drosophile et la souris s’effectue en deux étapes : la maturation du transcrit primaire et l’amplification, de type ping-pong [ 23] (➔). (➔)Voir la Synthèse de S. Muller etal.,m/s n° 5, mai 2013, page 487 Ces piARN de 22 à 30 nucléotides sont capables de différencier les séquences du soi de celles du non-soi et ainsi éliminer spécifiquement les « envahisseurs » [23]. Organisation génomique des piARN Dans les ovaires et testicules de drosophile, tout comme au stade pré-pachytène (troisième stade de la prophase) chez la souris, les piARN sont transcrits à partir de transposons et de séquences répétées qui en sont dérivées [25, 26]. Dans le génome, leurs gènes sont réunis en groupes de piARN, et la majorité d’entre eux sont monodirectionnels, ce qui signifie que tous les piARN d’un groupe sont produits à partir d’un seul des deux brins d’ADN. Maturation du piARN primaire Les précurseurs de piARN sont transcrits par l’ARN polymérase de type II et ne sont pas soumis à l’action de Dicer. La maturation des piARN primaires est un mécanisme peu connu. Initialement, un seul modèle, démontré chez la drosophile, décrivait succinctement ce mécanisme : le précurseur de piARN serait clivé par une endonucléase, Zucchini/ MitoPLD (mitochondrial phospholipase D) (non conservée chez C. elegans), qui marque, d’un groupement monophosphate, l’extrémité 5’ du piARN [27, 28] (Figure 3). Des expériences in vitro proposent que des protéines PIWI prennent en charge ce fragment d’ARN, assistées par une protéine chaperonne (Hsp90) [ 29] (Figure 3). Une exonucléase 3’-5’ (qui reste inconnue) couperait ensuite l’extrémité 3’ de ces précurseurs de piARN permettant ainsi à la méthyltransférase Hen1 (ou small RNA 2’-O-methyltransferase) d’ajouter un groupement méthyle à la position 2’ de l’extrémité 3’ [ 30], afin de former le piARN primaire mature, qui sera ainsi protégé de la dégradation. De récents travaux ont néanmoins montré que les piARN pouvaient être générés selon deux voies parallèles [ 31]. À côté de la voie décrite précédemment, les précurseurs de piARN peuvent également être clivés directement par les protéines Argonaute, Aub et Ago3, et voir leur extrémité 3’ réduite par Nibbler, une exonucléase 3’-5’ [31, 32]. La méthyltransférase Hen1 forme ensuite le piARN primaire mature, qui, associé à une protéine PIWI, constituera le piRISC. D’autres travaux ont révélé que les facteurs Armitage (Armi, une héli-case) et Yb (somatic piRNA biogenesis factor), impliqués dans la voie de biogénèse des piARN chez la drosophile, permettent, lorsqu’ils sont associés à un transcrit, de l’identifier comme précurseur de piARN [ 33], ce qui est essentiel à son engagement dans la voie de biogenèse. 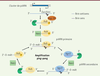 | Figure 3. Voie de biogenèse des piARN chez D. melanogaster. Le transcrit antisens (brin bleu) de piARN est clivé par Zucchini, qui génère l’extrémité 5’. Il est ensuite pris en charge par la protéine Aubergine (Aub). L’extrémité 3’ est alors raccourcie par l’action d’une exonucléase (qui reste inconnue) et finalement un groupement méthyle est ajouté à la position 2’ (2’-O-mét) du nucléotide en 3’ par Hen1. L’ensemble forme le piRISC qui va pouvoir cliver le brin sens (brin violet) de sa cible en un précurseur de piARN secondaire. Les piARN secondaires sens seront pris en charge par Ago3 et subiront une maturation par une exonucléase et Hen1. Ils pourront alors, à leur tour, cliver le brin antisens de leurs cibles en un pré-curseur de piARN antisens qui sera pris en charge, à nouveau, par Aub. |
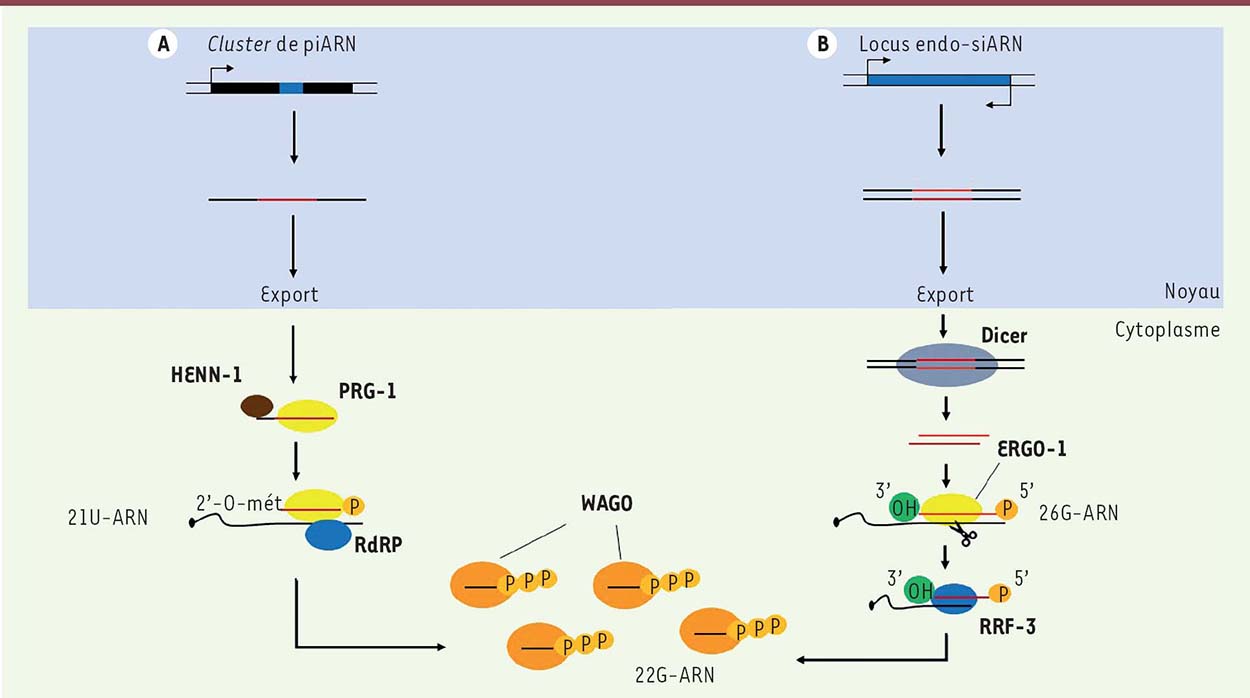
L’amplification ping-pong Le complexe effecteur piRISC peut cliver, entre les nucléotides 10 et 11 de son extrémité 5’, le brin sens de sa cible qui lui est complémentaire formant ainsi un nouveau piARN secondaire (Figure 3). Ce dernier sera alors pris en charge par une protéine PIWI, Ago3 chez la drosophile, ou MIWI2 chez la souris. L’ajout d’un groupement 2’-O-méthyle à l’extrémité 3’ formera le piARN secondaire mature. Il sera alors capable, à son tour, de cliver le brin antisens de sa cible, par complémentarité de bases. Ce cycle se poursuivra tant que les piARN secondaires reconnaîtront et cliveront des transposons afin de générer de grandes quantités de piARN pour permettre une répression efficace. La reconnaissance spécifique des brins antisens et sens par les protéines PIWI/Aub et Ago3 est un mécanisme qui reste encore inconnu. De nombreuses autre protéines interviennent dans la production des piARN, telles que les protéines de la famille Tudor et des hélicases à ARN [34]. Malheureusement, leurs fonctions moléculaires exactes dans la voie des piARN sont encore inconnues et des investigations supplémentaires reste nécessaires. Biogenèse des piARN chez C. elegans Chez le nématode, les piARN, aussi appelés 21U-ARN parce qu’ils sont constitués précisément de 21 nucléotides et qu’ils présentent une uridine à leur extrémité 5’, sont exprimés dans les cellules germinales [ 35]. Organisation génomique piARN 21U-ARN Les 21U-ARN sont codés dans les régions intergéniques et introniques de gènes codants une protéine [35, 36]. Chaque locus de 21U-ARN produit un court transcrit indépendant d’environ 26 nucléotides [ 35]. Maturation des 21U-ARN La maturation des 21U-ARN reste un processus peu connu. Les précurseurs de 21U-ARN sont transcrits par l’ARN polymérase de type II. Deux nucléotides en 5’ sont ensuite éliminés (par un mécanisme inconnu) afin de permettre leur prise en charge par une protéine Argonaute, PRG-1 (piwi-related gene 1) [ 37] (Figure 4). Le précurseur subit alors un raccourcissement de 3 nucléotides en 3’ (par un mécanisme qui reste inconnu) et une réaction de méthylation par HENN-1 (l’orthologue de Hen1) afin de former le 21U-ARN mature [ 37] (Figure 4). Une autre protéine Argonaute, homologue à PRG-1, PRG-2, a été décrite. Cependant, sa fonction n’a pas encore été clairement démontrée [ 38].  | Figure 4. Voie de biogenèse des piARN et siARN chez C. elegans. A. Le transcrit de piARN est exporté dans le cytoplasme pour pouvoir être pris en charge par la protéine Argonaute PRG-1 (piwi-related gene 1). HENN-1 forme alors le piARN primaire mature, aussi appelé 21U-ARN, en ajoutant un groupement méthyl à son extrémité 3’ (2’-O-mét). Le piRISC ainsi formé va reconnaître sa cible et recruter des RdRP (ARN polymérases ARN-dépendantes) qui vont produire des piARN secondaires, appelés 22G-ARN. Ces derniers, pour réprimer leurs cibles, s’associeront aux protéines WAGO (worm-specific Argonautes). B. Le long transcrit d’ARN double brin, après avoir été exporté dans le cytoplasme, est clivé par Dicer pour former un duplexe d’ARN. Les brins sont dissociés par un mécanisme encore inconnu et l’un des brins, appelé 26G-ARN, sera pris en charge par la protéine Argonaute ERGO-1. Le complexe effecteur ainsi formé va reconnaître son ARNm cible et le cliver. ERGO-1 recrute ensuite des RdRP qui utilisent l’ARNm comme modèle pour produire des siARN secondaires, appelés 22G-ARN. Ces derniers vont être pris en charge par les protéines WAGO (telles que HRDE-1 [heritable RNAi deficient 1] et NRDE-3[nuclear RNAi defective 3]) et ainsi réprimer leurs cibles. |
Production des 22G-ARN Les 21U-ARN complexés à PRG-1 s’associent à leurs cibles en tolérant quelques mésappariements de bases, et recrutent des ARN polymérases ARN-dépendantes ou RdRP (RNA-dependent RNA polymerases) (Figure 4). Ces dernières entraînent la production massive de siARN secondaires (ou 22G-ARN), des ARN de 22 nucléotides possédant un groupement triphosphate à leur extrémité 5’. Ces 22G-ARN sont pris en charge par des protéines WAGO (worm-specific Agos) (Figure 4), dont la protéine Argonaute HRDE-1 (heritable RNAi defective 1, ou WAGO-9), dans les cellules germinales, et NRDE-3 (nuclear RNAi defective 3, ou WAGO-12), dans les cellules somatiques, afin de réprimer l’expression de gènes envahisseurs de manière transcriptionnelle ou post-transcription-nelle. Une voie de protection du soi, qui peut entrer en compétition avec la voie de répression reposant sur le complexe WAGO/22G-ARN, est activée lorsque les 22G-ARN sont pris en charge par une autre protéine Argonaute, CSR-1 [ 39]. Le rôle exact de cette protéine dans la voie des ARN courts non codants reste controversé et fait l’objet de nombreuses études. Comme chez la drosophile et la souris, d’autres protéines sont nécessaires pour la production de piARN, comme les protéines TOFU (twenty-one-u fouled ups), la protéine PID-1 (piRNA-induced silencing defective-1) et la protéine PRDE-1 (piRNA silencing defective 1) [40-42]. À ce jour, leur fonction dans la voie des piARN est encore mal comprise et demande plus d’investigations. |
La voie de biogenèse des siARN endogènes Les petits ARN interférents, communément appelés siARN, sont de courts ARN non codants qui régulent l’expression des gènes en provoquant la dégradation de l’ARNm. Chez C. elegans, il existe deux voies de biogenèse des siARN : la voie exogène et la voie endogène. Les siARN exogènes sont produits après introduction dans la cellule d’ARN double brin exogène. Ils sont à l’origine de l’ARNi (interférence à l’ARN), un mécanisme mis en évidence par Fire et Mello en 1998 [43]. Nous nous intéresserons essentiellement aux siARN endogènes chez C. elegans. Les siARN endogènes sont bien conservés au cours de l’évolution, de C. elegans et D. melanogaster jusqu’aux mammifères. Nous ne décrirons cependant leur voie de biogénèse que chez le nématode, celle-ci restant inconnue chez la drosophile et les mammifères. La voie de biogenèse des siARN endogènes présente quelques similarités avec celle des miARN. Leur mode de régulation, quant à lui, se rapproche de celui des piARN puisqu’ils ciblent principalement des éléments transposables. Maturation des siARN endogènes primaires Les siARN endogènes sont transcrits à partir de séquences codantes du génome. Un long ARN double brin est clivé par Dicer pour former un duplex d’ARN long de 26 nucléotides présentant un groupement monophosphate en 5’, un groupe hydroxyle en 3’ et deux nucléotides flottants en 3’ [ 44] (Figure 4). De façon similaire aux miARN, les siARN primaires, aussi appelées 26G-ARN, vont être pris en charge par une protéine Argonaute appelée ERGO-1 [ 45] (Figure 4). La dissociation des deux brins d’ARN, par un mécanisme encore inconnu, conduira à la formation du RISC. Production des siARN secondaires endogènes Le complexe effecteur RISC reconnaît avec une complémentarité parfaite la cible qu’il clive. L’Argonaute ERGO-1 recrute alors une RdRP, RRF-3, qui utilisera l’ARNm cible comme modèle afin de produire des siARN secondaires [ 46] (Figure 4). Ces siARN secondaires, plus abondants que les siARN primaires, sont appelées 22G-siARN. Ils permettent de renforcer le processus de dégradation de l’ARNm. De polarité antisens par rapport à l’ARNm, initialement ciblés par des 21U-ARN, ils s’associent ensuite aux protéines Argonautes WAGO (au nombre de 18) [ 47], pour réprimer efficacement l’expression du gène ciblé. Dans les gonades mâles, les 26G-ARN sont pris en charge par les protéines Argonaute ALG-3 et ALG-4 afin d’initier la production de 22G-ARN par des RdRP qui, une fois associés aux WAGO, réguleront la spermatogénèse [ 48]. Chez les mammifères et la drosophile, il n’existe pas de RdRP. Cependant, il a été montré, chez la souris et la drosophile, que, malgré l’absence de ces protéines, une production naturelle de siARN endogènes fonctionnels était réalisée à partir de molécules d’ARN double brin [49-52]. Ces derniers aideraient les piARN à réguler l’expression des rétrotransposons mais aussi les miARN à réguler les ARNm [53]. |
Chaque catégorie d’ARN courts non codants possède une voie de biogénèse qui lui est propre, mais certaines voies peuvent partager des facteurs communs. Ainsi, les miARN et les siARN sont clivés par une seule et même enzyme, Dicer, chez C. elegans et les mammifères, alors que deux enzymes Dicer distinctes sont utilisées chez la drosophile. De même, chez C. elegans, la voie de biogénèse des piARN fusionne avec celle des siARN pour former des 22G-ARN. Les ARN courts non codants, aussi appelés micromanagers, ont un puissant potentiel de répression et sont très conservés au cours de l’évolution, suggérant un rôle important dans la cellule. Ils sont impliqués dans de nombreux processus biologiques tels que le développement, l’apoptose, la différenciation et la prolifération cellulaires, la spermatogenèse, la ségrégation des chromosomes et la défense contre des « envahisseurs » du génome. Ceci fait donc de ces petits ARN des molécules essentielles au bon fonctionnement de la cellule. Ce nouveau domaine de recherche suscite beaucoup d’intérêts; toutefois, il reste encore de nombreux aspects de la biologie des ARN courts non codants à étudier et comprendre afin d’éclairer la biologie du vivant. 0 |
Nous tenons à remercier les membres du groupe de recherche pour leurs commentaires et suggestions. Ce travail a été supporté par le soutien financier du Conseil de recherches en sciences naturelles et génie du Canada et des Instituts de recherche en santé du Canada. M.J.S. est récipiendaire d’une chaire de recherche des Fonds de recherche du Québec-Santé. Restreints dans le nombre de références, nous nous excusons auprès de tous ceux et celles que nous n’avons pas cité. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article. |
Footnotes |
1. Lee RC, Feinbaum RL, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. 1993;; 75 : :843.-54. 2. Romero Y, Calvel P, Nef S. Petits ARN non codants et spermatogenèse. Med Sci (Paris). 2012;; 28 : :490.-6. 3. Rie D de, Abugessaisa I, Alam T, et al. An integrated expression atlas of miRNAs and their promoters in human and mouse. Nat Biotechnol. 2017;; 35 : :872.-8. 4. Griffiths-Jones S, Saini HK, Dongen S Van, et al. miRBase: Tools for microRNA genomics. Nucleic Acids Res. 2008;; 36 : :D154.-8. 5. Saini HK, Griffiths-Jones S, Enright AJ. Genomic analysis of human microRNA transcripts. Proc Natl Acad Sci USA. 2007;; 104 : :17719.-24. 6. Ozsolak F, Poling LL, Wang Z, et al. Chromatin structure analyses identify miRNA promoters. Genes Dev. 2008;; 22 : :3172.-83. 7. Fujita S, Iba H. Putative promoter regions of miRNA genes involved in evolutionarily conserved regulatory systems among vertebrates. Bioinformatics. 2008;; 24 : :303.-8. 8. Corcoran DL, Pandit K V., Gordon B, et al. Features of mammalian microRNA promoters emerge from polymerase II chromatin immunoprecipitation data. PLoS One. 2009;; 4 : :e5279.. 9. Chien CH, Sun YM, Chang WC, et al. Identifying transcriptional start sites of human microRNAs based on high-throughput sequencing data. Nucleic Acids Res. 2011;; 39 : :9345.-56. 10. Nguyen TA, Jo MH, Choi Y-G, et al. Functional anatomy of the human microprocessor. Cell. 2015;; 161 : :1374.-87. 11. Lund E, Güttinger S, Calado A, et al. Nuclear export of microRNA precursors. Science. 2004;; 303 : :95.-8. 12. Kim YK, Kim B, Kim VN. Re-evaluation of the roles of DROSHA, Exportin 5, and DICER in microRNA biogenesis . Proc Natl Acad Sci USA. 2016;; 113 : :E1881.-9. 13. Diederichs S, Haber DA. Dual role for Argonautes in microRNA processing and posttranscriptional regulation of microRNA expression . Cell 2007;; 131 : :1097.-108. 14. Bouasker S, Simard MJ. The slicing activity of miRNA-specific Argonautes is essential for the miRNA pathway. in C. elegans. Nucleic Acids Res. 2012;; 40 : :10452.-62. 15. Hutvagner G, Simard MJ. Argonaute proteins: key players in RNA silencing. Nat Rev Mol Cell Biol. 2008;; 9 : :22.-32. 16. Iwasaki S, Kobayashi M, Yoda M, et al. Hsc70/Hsp90 chaperone machinery mediates ATP-dependent RISC loading of small RNA duplexes. Mol Cell. 2010;; 39 : :292.-9. 17. Iwasaki S, Sasaki HM, Sakaguchi Y, et al. Defining fundamental steps in the assembly of the Drosophila RNAi enzyme complex. Nature. 2015;; 521 : :533.-6. 18. Schwarz DS, Hutvágner G, Du T, et al. Asymmetry in the assembly of the RNAi enzyme complex . Cell 2003;; 115 : :199.-208. 19. Khvorova A, Reynolds A, Jayasena SD. Functional siRNAs and miRNAs exhibit strand bias. Cell 2003;; 115 : :209.-16. 20. Chung WJ, Agius P, Westholm JO, et al. Computational and experimental identification of mirtrons in Drosophila melanogaster and Caenorhabditis elegans. Genome Res. 2011;; 21 : :286.-300. 21. Cheloufi S, Santos CO Dos, Chong MMW, et al. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis. Nature. 2010;; 465 : :584.-9. 22. Cifuentes D, Xue H, Taylor DW, et al. A novel miRNA processing pathway independent of Dicer requires Argonaute2 catalytic activity. Science. 2010;; 328 : :1694.-8. 23. Muller S, Pandey RR, Pillai RS. Les piARN forgent un système immunitaire pour le génome. Med Sci (Paris). 2013;; 29 : :487.-94. 24. Lim RSM, Kai T. A piece of the pi(e): the diverse roles of animal piRNAs and their PIWI partners. Semin Cell Dev Biol. 2015;; 47-48 : :17.-31. 25. Watanabe T, Takeda A, Tsukiyama T, et al. Identification and characterization of two novel classes of small RNAs in the mouse germline. : Retrotransposon-derived siRNAs in oocytes and germline small RNAs in testes. Genes Dev. 2006;; 20 : :1732.-43. 26. Girard A, Sachidanandam R, Hannon GJ, et al. A germline-specific class of small RNAs binds mammalian Piwi proteins. Nature. 2006;; 442 : :199.-202. 27. Nishimasu H, Ishizu H, Saito K, et al. Structure and function of Zucchini endoribonuclease in piRNA biogenesis. Nature. 2012;; 491 : :284.-7. 28. Ipsaro JJ, Haase AD, Knott SR, et al. The structural biochemistry of Zucchini implicates it as a nuclease in piRNA biogenesis. Nature. 2012;; 491 : :279.-83. 29. Kawaoka S, Izumi N, Katsuma S, et al. 3’ End formation of PIWI-interacting RNAs in vitro. Mol Cell. 2011;; 43 : :1015.-22. 30. Horwich MD, Li C, Matranga C, et al. The Drosophila RNA methyltransferase, DmHen1, modifies germline piRNAs and single-stranded siRNAs in RISC. Curr Biol. 2007;; 17 : :1265.-72. 31. Hayashi R, Schnabl J, Handler D, et al. Genetic and mechanistic diversity of piRNA 3’-end formation. Nature. 2016;; 539 : :588.-92. 32. Feltzin VL, Khaladkar M, Abe M, et al. The exonuclease Nibbler regulates age-associated traits and modulates piRNA length in Drosophila. Aging Cell. 2015;; 14 : :443.-52. 33. Pandey RR, Homolka D, Chen K-M, et al. Recruitment of Armitage and Yb to a transcript triggers its phased processing into primary piRNAs in Drosophila ovaries. PLOS Genet. 2017;; 13 : e1006956.. 34. Siomi MC, Sato K, Pezic D, et al. PIWI-interacting small RNAs: the vanguard of genome defence. Nat Rev Mol Cell Biol. 2011;; 12 : :246.-58. 35. Ruby JG, Jan C, Player C, et al. Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs. in C. elegans. Cell. 2006;; 127 : :1193.-207. 36. Batista PJ, Ruby G, Claycomb JM, et al. PRG-1 and 21U-RNAs interact to form the piRNA complex required for fertility in C. elegans. Mol Cell. 2008;; 31 : :67.-78. 37. Montgomery TA, Rim YS, Zhang C, et al. PIWI associated siRNAs and piRNAs specifically require the Caenorhabditis elegans HEN1 ortholog henn-1. PLoS Genet . 2012;; 8 : e1002616.. 38. Wang G, Reinke V. A C. elegans Piwi, PRG-1, Regulates 21U-RNAs during spermatogenesis. Curr Biol. 2008;; 18 : :861.-7. 39. Wedeles C, Wu M, Claycomb J. Protection of germline gene expression by the C. elegans argonaute CSR-1. Dev Cell. 2013;; 27 : :664.-71. 40. Goh WSS, Seah JWE, Harrison EJ, et al. A genome-wide RNAi screen identifies factors required for distinct stages of C. elegans piRNA biogenesis. Genes Dev. 2014;; 28 : :797.-807. 41. Albuquerque BFM de, Luteijn MJ, Cordeiro Rodrigues RJ, et al. PID-1 is a novel factor that operates during 21U-RNA biogenesis in Caenorhabditis elegans. Genes Dev. 2014;; 28 : :683.-8. 42. Weick EM, Sarkies P, Silva N, et al. PRDE-1 is a nuclear factor essential for the biogenesis of Ruby motif-dependent piRNAs in C. elegans. Genes Dev. 2014;; 28 : :783.-96. 43. Fire A, Xu S, Montgomery MK, et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 1998;; 391 : :806.-11. 44. Ketting RF, Fischer SEJ, Bernstein E, et al. Dicer functions in RNA interference and in synthesis of small RNA involved in developmental timing in C. elegans. Genes Dev. 2001;; 15 : :2654.-9. 45. Gent JI, Lamm AT, Pavelec DM, et al. Distinct phases of siRNA synthesis in an endogenous RNAi pathway in C. elegans soma. Mol Cell. 2010;; 37 : :679.-89. 46. Han T, Manoharan AP, Harkins TT, et al. 26G endo-siRNAs regulate spermatogenic and zygotic gene expression in Caenorhabditis elegans. Proc Natl Acad Sci USA. 2009;; 106 : :18674.-9. 47. Yigit E, Batista PJ, Bei Y, et al. Analysis of the C. elegans Argonaute family reveals that distinct Argonautes act sequentially during RNAi. Cell. 2006;; 127 : :747.-57. 48. Conine CC, Batista PJ, Gu W, et al. Argonautes ALG-3 and ALG-4 are required for spermatogenesis-specific 26G-RNAs and thermotolerant sperm in Caenorhabditis elegans . Proc Natl Acad Sci USA. 2010;; 107 : :3588.-93. 49. Czech B, Malone CD, Zhou R, et al. An endogenous small interfering RNA pathway in Drosophila. Nature. 2008;; 453 : :798.-802. 50. Okamura K, Chung WJ, Ruby JG, et al. The Drosophila hairpin RNA pathway generates endogenous short interfering RNAs. Nature. 2008;; 453 : :803.-6. 51. Tam OH, Aravin AA, Stein P, et al. Pseudogene-derived small interfering RNAs regulate gene expression in mouse oocytes. Nature. 2008;; 453 : :534.-8. 52. Watanabe T, Totoki Y, Toyoda A, et al. Endogenous siRNAs from naturally formed dsRNAs regulate transcripts in mouse oocytes. Nature. 2008;; 453 : :539.-43. 53. Piatek MJ, Werner A. Endogenous siRNAs, regulators of internal affairs. Biochem Soc Trans. 2014;; 42 : :1174.-9. 54. Bohnsack MT, Czaplinski K, Gorlich D. Exportin 5 is a RanGTP-dependent dsRNA-binding protein that mediates nuclear export of pre-miRNAs. RNA. 2004;; 10 ::185.-91. 55. Yi R, Qin Y, Macara IG, Cullen BR. Exportin-5 mediates the nuclear export of pre-microRNAs and short hairpin RNAs. Genes Dev. 2003;; 17 : :3011.-6. 56. Büssing I, Yang JS, Lai EC, Grosshans H. The nuclear export receptor XPO-1 supports primary miRNA processing in C. elegans and Drosophila. EMBO J. 2010;; 29 : :1830.-9. |