Au cours d’une infection virale, le virus, « parasite » intracellulaire obligatoire, se multiplie au sein des cellules hôtes. Il libère son génome dans la cellule hôte et détourne les machineries moléculaires de celle-ci afin de se répliquer et de produire de nouvelles particules virales, ou virions, qui infecteront, par la suite, d’autres cellules. Certaines cellules ont cependant la capacité d’interférer avec la réplication virale en exprimant des protéines jouant le rôle de facteurs de restriction. L’expression de ces facteurs est constitutive mais pour la plupart d’entre eux, elle est accrue au cours de la réponse immunitaire antivirale innée. La reconnaissance de motifs viraux par des récepteurs cellulaires de l’immunité innée (ou senseurs) induit, en effet, une réponse interféron de type I qui, ellemême, gouverne l’expression de nombreux facteurs de restriction.
Une grande diversité de facteurs de restriction a été décrite. Ils agissent sur différents composants du virus comme le génome ou les protéines virales. Ces facteurs peuvent agir sur un virus en particulier, ou sur différentes familles de virus. Par exemple, les facteurs APOBEC3G (apolipoprotein B mRNA-editing, enzyme-catalytic, polypeptide-like 3G) et SAMHD1 (sterile alpha motif and HD domain 1) inhibent la transcription inverse du rétrovirus VIH-1 (virus de l’immunodéficience humaine de type 1) en causant, respectivement, des mutations de glycine en alanine lors de la transcription inverse [1], ou une diminution de la quantité de dNTP (désoxynucléotides triphosphates) cellulaires disponibles [2]. Le facteur MxA (myxovirus resistance gene A) agit, quant à lui, sur un large spectre de virus à ARN négatif (comme le bunyavirus, l’hantavirus, l’orthomyxovirus, le paramyxovirus, le rhabdovirus et l’alphavirus) en ciblant les nucléoprotéines qui permettent l’encapsidation du génome viral assurant la réplication et la transcription du virus; MxA, en formant des oligomères entourant les nucléoprotéines virales, inhibe ces fonctions [3, 4].
Les protéines transmembranaires IFITM (interferon-induced transmembrane protein), que nous décrirons par la suite, exercent un effet inhibiteur sur différents virus enveloppés (c’est-à-dire délimités par une bicouche lipidique dérivée des membranes cellulaires) issus de familles diverses telles les orthomyxovirus, les rétrovirus, les filovirus, les coronavirus, etc. [5](➔).
Ces virus se sont cependant adaptés au cours de l’évolution et ont développé des mécanismes qui leur permettent d’outrepasser ces facteurs de restriction. Ainsi, le virus VIH-1 exprime des protéines dites auxiliaires, comme Vif (viral infectivity factor) qui inhibe APOBEC3G. De même, les rétrovirus VIH-2 (virus de l’immunodéficience humaine de type 2) et SIVMAC (virus de l’immunodéficience simienne du macaque) produisent la protéine Vpx (viral protein x) qui inhibe SAMHD1.
(➔) Voir la Synthèse de K. Tatour et A. Cimarelli, m/s n° 4, avril 2015, page 377
Chez l’homme, trois membres de la famille des IFITM assurent cette action antivirale : les IFITM-1, -2 et -3. Une première série d’études a mis en évidence le mécanisme par lequel ces IFITM peuvent interférer avec la réplication des virus enveloppés. Les IFITM, présents dans la membrane cellulaire, préviennent la libération du virus dans le cytoplasme en inhibant l’étape de fusion entre membranes virales et cellulaires. Des études récentes ont cependant identifié, dans le cas du rétrovirus VIH-1, un mécanisme antiviral supplémentaire pour les facteurs IFITM, notamment pour IFITM-3. IFITM-3 est en effet incorporée dans l’enveloppe des virions, engendrant ainsi une réduction de la capacité fusogène des particules virales et donc de leur pouvoir infectieux [6-8]. Le virus VIH-1 peut toutefois acquérir une résistance à l’action de l’IFITM-3 par des variations de séquence de sa glycoprotéine d’enveloppe gp120 [9].
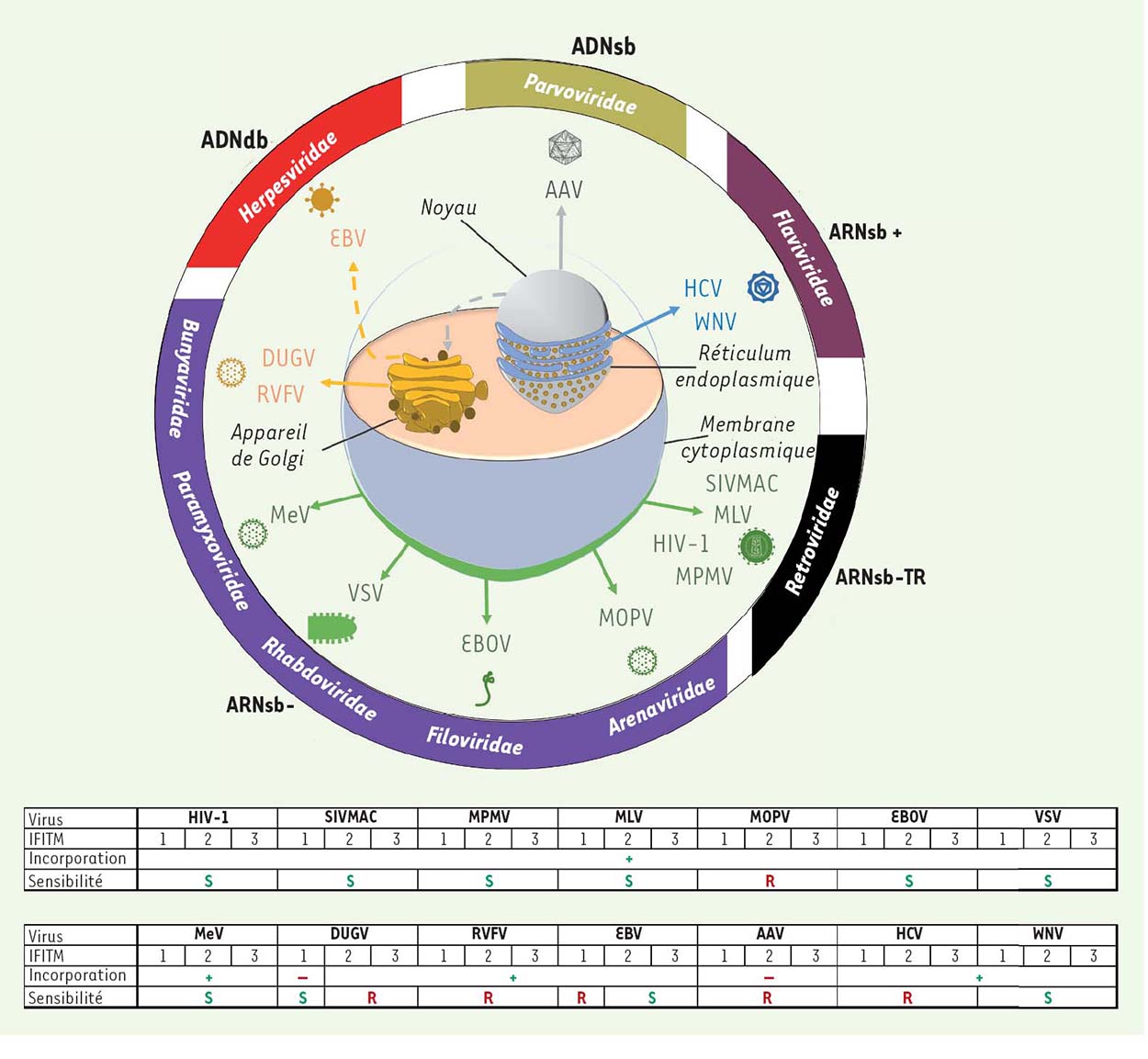
Se fondant sur ces données, une étude récente collaborative menée par les équipes du centre international de recherche en infectiologie de Lyon (CIRI, U1111), de Tours (U966) et de Paris (UMR955) a cherché à déterminer si la réduction du pouvoir infectieux des virions par les IFITM, identifiée dans le cas du VIH-1, pouvait être conservée et rester efficace sur un panel de virus différant en termes de génome, de cycle de réplication et de mode d’assemblage des virions (Figure 1) [10]. Pour cela, l’effet des IFITM a été examiné sur le pouvoir infectieux de quatorze virus regroupant des virus à ARN négatif (le virus de la stomatite vésiculaire, VSV; le virus de la rougeole, MeV; le virus Ebola, EBOV; le virus Dugbe, DUGV; le virus de la fièvre de la vallée du Rift, RVFV et le virus Mopeia, MOPV), des virus à ARN positif (le virus West Nile, WNV et le virus de l’hépatite C, HCV), des rétrovirus (le virus de l’immunodéficience simienne du macaque, SIVMAC; le virus de la leucémie murine, MLV; le virus simien Mason-Pfizer, MPMV) et des virus à ADN (le virus Epstein-Barr, EBV et le virus adéno-associé, AAV). Le rétrovirus VIH-1 a été utilisé comme contrôle de l’action anti-infectieuse des IFITM dans cette étude. Notons que l’ensemble de ces virus, à l’exception d’AAV, possède une enveloppe, ce qui pourrait moduler l’action des IFITM sur ces virus.
| Figure 1 Mode d’assemblage des virions et influence des protéines IFITM sur le pouvoir infectieux des particules virales néoformées. Les différents virus sont représentés avec leur caractéristique génomique et leur groupe d’appartenance. ARNsb - : génome à ARN simple brin de polarité néga t i v e; ARNsb-TR : génome à ARN simple brin et nécessitant une transcriptase inverse; ARNsb + : génome à ARN simple brin de polarité positive; ADNsb : génome à ADN simple brin; ADNdb : génome à ADN double brin. Les virus étudiés sont indiqués. VIH-1 : virus de l’immunodéficience humaine de type 1; SIVMAC : virus de l’immunodéficience simienne du macaque; MPMV : virus simien Mason-Pfizer; MLV : virus de la leucémie murine; MOPV : virus Mopeia; EBOV : virus Ebola; VSV : virus de la stomatite vésiculaire; MeV : virus de la rougeole; DUGV : virus Dugbe; RVFV : virus de la fièvre de la vallée du Rift; EBV : virus Epstein-Barr; AAV : virus adéno-associé; HCV : virus de l’hépatite C; WNV : virus West Nile. Les tableaux récapitulent le rôle des IFITM pour chaque virus, à savoir leur incorporation dans les particules virales (indiquée par + ou -); La résistance ou la sensibilité des virus aux IFITM sont notées respectivement par R et S (adapté de [10]). |
Afin de déterminer l’influence des IFITM sur l’ensemble de ces virus, les particules virales ont été produites dans des cellules qui expriment ou n’expriment pas ces protéines. Le génome viral a été cloné puis éventuellement co-transfecté avec les gènes codant les IFITM. Alternativement, les cellules ont été infectées après transfection d’un IFITM particulier dans des conditions qui s’affranchissent d’une inhibition de l’entrée virale par les IFITM, ce qui permet d’analyser leur impact uniquement sur la production des particules virales infectieuses. Les virions produits dans les cellules exprimant ou non les IFITM ont été ensuite purifiés et quantifiés. À noter que la présence des IFITM n’influence pas la quantité de particules virales produites. L’incorporation des IFITM dans les virions a été détectée par analyse protéique (par western blot) pour l’ensemble des virus de l’étude (à l’exception du virus DUGV). Aucune incorporation n’a été détectée pour le virus AAV non-enveloppé. L’association d’IFITM aux virions a été confirmée par microscopie électronique pour sept virus (SIVMAC, MLV, MPMV, VSV, MeV et EBV).
Une interaction entre les glycoprotéines d’enveloppe du VIH-1 et les protéines IFITM-1, -2 et -3, conduisant à une diminution de l’incorporation des glycoprotéines virales lors de l’assemblage des virions avait été montrée [8]. Contrastant avec ces résultats concernant le VIH-1, aucune diminution de l’incorporation des glycoprotéines virales dans les particules n’a été observée avec les virus enveloppés SIVMAC, MLV, MPMV, VSV, EBOV, MeV et RVFV.
Une diminution du pouvoir infectieux des virions produits par les cellules exprimant les IFITM a été confirmée pour les rétrovirus VIH-1 et SIVMAC (Figure 1). Cette inhibition a également été observée pour d’autres virus comme MLV, MPMV, VSV, MeV, EBOV et WNV. Pour les virus VSV, MeV et EBOV, les résultats des expériences d’inhibition de l’expression des facteurs IFITM endogènes (par knockdown) ont confirmé l’effet antiviral des IFITM. Certains virus ne sont cependant pas sensibles à l’action des trois IFITM, mais seulement à l’un ou plusieurs d’entre eux. Les virus HCV, RVFV, MOPV et AAV ne sont par contre pas sensibles à l’influence des IFITM. Il faut toutefois rappeler que la résistance d’AAV aux IFITM n’est pas surprenante du fait de l’absence d’enveloppe virale.
L’incorporation des IFITM dans les virions ne prédit en fait pas un effet antiviral. En effet, les virus qui résistent aux IFITM (à l’exception d’AAV) incorporent également ces protéines dans leur propre enveloppe virale, suggérant que la présence de ces protéines dans les particules néoformées n’est pas suffisante pour réduire le pouvoir infectieux des virions. Inversement, un effet antiviral lié aux IFITM peut s’exercer en l’absence même d’incorporation d’IFITM dans les particules, comme dans le cas du virus DUGV en présence d’IFITM-1. Les IFITM pourraient donc laisser une « empreinte négative » sur les particules virales lors de leur assemblage dans les cellules productrices, et cela indépendamment de leur incorporation dans les particules virales.
Des études antérieures avaient montré, dans le cas du rétrovirus VIH-1, que les glycoprotéines d’enveloppe virale pouvaient être impliquées dans la sensibilité des virions aux IFITM [9, 11, 12]. Le mode d’assemblage des virions pourrait ainsi moduler l’effet antiviral des IFITM. Afin d’identifier le facteur déterminant la sensibilité des virus aux IFITM, des permutations génétiques ont été réalisées entre les séquences nucléotidiques des capsides des virus sensibles (VIH-1 et VSV) et celles des glycoprotéines d’enveloppe des virus résistants (HCV et RVFV), produisant ainsi des virus pseudotypés. Le pouvoir infectieux des virions a été évalué et les résultats obtenus suggèrent que la nature de la capside, donc le mode d’assemblage des virions, et non celle de la glycoprotéine d’enveloppe, détermine la sensibilité d’un virus aux protéines IFITM.
Les protéines IFITM agissent sur le pouvoir infectieux des particules virales néoformées mais elles protègent également les cellules d’une nouvelle infection. Il était donc intéressant d’examiner si ces deux mécanismes coexistaient au sein d’une même cellule infectée. Pour cela, des cellules exprimant la protéine IFITM-3 de façon inductible ont été infectées par différents virus et la capacité d’IFITM-3 à protéger les cellules a été évaluée peu après l’infection afin d’analyser l’efficacité du premier cycle de la réplication virale. En parallèle, des infections à long terme ont été réalisées afin d’étudier le pouvoir infectieux des virions nouvellement produits par la cellule en présence ou en l’absence d’expression d’IFITM-3. Ces deux analyses montrent à la fois une protection des cellules cibles en présence d’IFITM-3, et une réduction du pouvoir infectieux des virions produits, et cela pour des virus aussi différents que le VIH-1 et l’EBOV.
L’IFITM reste actuellement le seul facteur connu pour présenter une capacité d’altérer le cycle viral à deux étapes distinctes. Cette inhibition bimodale du pouvoir infectieux des virus par les protéines IFITM, lors de l’entrée virale, d’une part, et lors de la production de virions, d’autre part, est conservée pour un très large panel de virus. Pourtant, peu de données existent concernant les partenaires cellulaires d’IFITM qui participeraient à cette activité antivirale. Des travaux se sont intéressés aux cofacteurs cellulaires qui pourraient participer à l’activité antivirale des protéines IFITM. Lors de l’entrée virale, IFITM-3 pourrait ainsi interagir avec la protéine VAPA (vesicle-associated membrane protein [VAMP]-associated protein A) dans les endosomes et inhiber son interaction avec OSBP (oxysterolbinding protein). Ceci affecterait la régulation intracellulaire du cholestérol, engendrant son accumulation dans la membrane de l’endosome, ce qui interférerait avec la fusion des membranes virales et cellulaires [13]. Les souris déficientes pour la protéine ZMPSTE24 (zinc metallopeptidase STE24), une métalloprotéase transmembranaire qui peut être co-immunoprécipitée avec les IFITM, présentent, après infection par le virus Influenza, une charge virale plus importante et un taux de mortalité plus élevé que les souris sauvages [14], suggérant que ZMPSTE24 pourrait contribuer à l’activité antivirale des IFITM.
Les rôles des cofacteurs cellulaires dans l’activité antivirale des IFITM restent donc à étudier. Nous pouvons cependant concevoir que les modifications des membranes endosomales ou virales pourraient interférer avec les cycles de réplication d’un grand nombre de virus enveloppés.
The double antiviral effect of IFITM is conserved in many species of enveloped viruses