Enterococcus faecalis est une bactérie naturellement présente dans le microbiote gastro-intestinal chez l’homme [ 1], qui peut être à l’origine d’infections nosocomiales opportunistes lorsqu’elle colonise des plaies ou le système sanguin [ 2]. Les souches d’E. faecalis les plus pathogènes sont celles qui ont acquis une multirésistance aux antibiotiques (à la vancomycine, au linézolide, etc.) [ 3], rendant le traitement de ces infections particulièrement difficile. Cette multi-résistance est acquise par transfert horizontal d’éléments génétiques mobiles (MGE, mobile genetic elements) portant des gènes de résistance [ 4, 5].
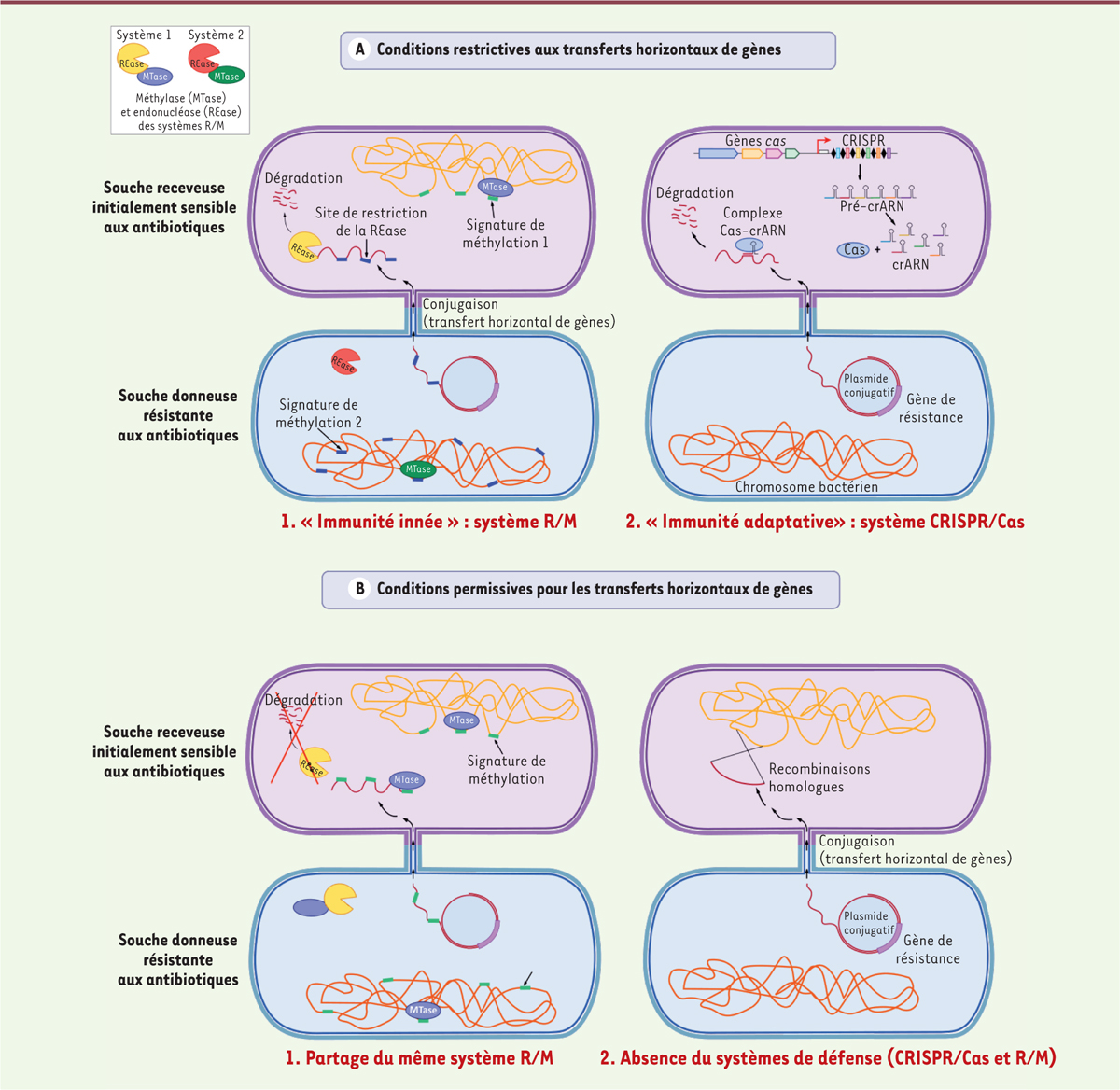
Néanmoins, les bactéries possèdent des systèmes de défense contre les éléments génétiques mobiles : le système restriction/modification (R/M) et le système CRISPR/Cas, qui sont souvent présentés comme des mécanismes bactériens d’« immunité innée » et d’« immunité adaptative », respectivement. Le système R/M repose sur le clivage non spécifique du matériel génétique entrant par une endonucléase (fonction R, restriction, du système R/M), et sur la protection du génome bactérien par méthylation (action de la méthylase M associée). La signature de méthylation étant propre à chaque système R/M, ce système permet donc la distinction du soi et du non-soi. Les systèmes CRISPR/Cas sont, quant à eux, fondés sur une reconnaissance spécifique de séquences génétiques étrangères à l’aide d’ARN guides (transcrits à partir des séquences espaceurs du CRISPR) associés à des endonucléases Cas. À la différence des systèmes R/M, ils possèdent une « mémoire » des précédentes rencontres avec des éléments génétiques étrangers ( Figure 1 ).
| Figure 1.
Les limites des mécanismes de défense bactériens face aux transferts horizontaux de gènes. Les bactéries, dont E. faecalis, disposent de systèmes de défense endogènes leur permettant de lutter contre les transferts horizontaux de gènes et ainsi protéger leur propre génome. Elles possèdent ainsi un système de restriction-modification (R/M) (A.1.) reposant sur la protection du génome endogène par méthylation spécifique de site via la méthylase MTase (modification) et sur la dégradation des éléments génétiques exogènes par des endonucléases REase (restriction). Le second système, nommé CRISPR/Cas (A.2.), repose sur la reconnaissance spécifique de séquence d’un matériel génétique étranger entrant, par des endonucléases Cas associées à des ARN guides, les crARN, codés par les séquences espaceurs du locus CRISPR. Dans ces deux cas - A.1. et A.2. -, la bactérie receveuse n’acquiert pas de résistance aux antibiotiques, même après le processus de conjugaison, car les gènes de résistance transmis par la souche donneuse sont dégradés. Cependant, des déficiences de ces systèmes de défense peuvent conduire à la facilitation des transferts horizontaux de gènes. Le premier cas de figure est le partage d’un même système R/M entre la bactérie donneuse et receveuse, associé à l’absence de reconnaissance du matériel génétique conjugatif par le système CRISPR/Cas de la bactérie receveuse (B.1.). L’élément génétique transmis sera alors reconnu comme du « soi » par la bactérie receveuse, d’après la signature de méthylation qu’il porte, et ne sera dégradé ni par le système CRISPR/Cas, ni par le système R/M. Le second cas correspond à l’absence de systèmes de défense (R/M et CRISPR/Cas) fonctionnels (B.2.), ce qui entraîne l’acceptation de l’élément génétique étranger entrant par la bactérie receveuse et sa conservation. Dans ces deux conditions - B.1. et B.2. -, la bactérie receveuse, initialement sensible aux antibiotiques, acquiert un gène de résistance par ce transfert horizontal de gènes et devient elle-même résistante aux antibiotiques au même titre que la bactérie donneuse. |
Ces deux systèmes sont naturellement présents chez E. faecalis, limitant ainsi les transferts horizontaux de gènes. Ainsi, il est intéressant d’étudier les mécanismes ayant permis l’acquisition de résistances aux antibiotiques chez E. faecalis. De précédentes études génomiques ont révélé une perte des systèmes de défense contre les éléments génétiques étrangers chez les souches E. faecalis pathogènes résistantes aux antibiotiques [ 6], avec néanmoins la conservation du segment CRISPR2 dit « orphelin » car non associé à des gènes cas. L’hypothèse de Price et al. [ 7] est que l’absence ou la non-fonctionnalité de ces systèmes de défense chez une souche d’E. faecalis la rendraient « immunodéprimée » et donc incapable de se défendre contre les transferts horizontaux, ce qui permettrait alors l’acquisition des multirésistances.
Dans ce contexte, les auteurs ont cherché à comprendre l’impact de ces mécanismes de défense sur les transferts horizontaux de type conjugatif. En effet, la présence de plasmides conjugatifs à spectre d’hôtes étroit (plasmides PRP, pheromone-responsive plasmids), associés à une dissémination rapide de l’antibiorésistance, est décrite chez E. faecalis. Les auteurs ont ainsi testé le transfert horizontal d’un plasmide conjugatif de type PRP entre des souches d’E. faecalis donneuses et receveuses. La souche receveuse utilisée, nommée T11, est très proche génétiquement d’une souche pathogène multirésistante mais, contrairement à cette dernière, elle possède un système R/M (EfaRF1) et un système CRISPR3/Cas fonctionnels et n’est pas multirésistante aux antibiotiques. La souche donneuse, nommée OG1RF, possède un système R/M identique à celui de T11. Dans le cas de la souche receveuse T11, le plasmide conjugatif est reconnu à la fois par le système R/M et par CRISPR/Cas. Dans le cas de la souche receveuse T11 ∆cas9 (ne possédant pas de système CRISPR/Cas fonctionnel), le plasmide peut être reconnu par le système R/M commun aux deux souches mais n’est pas reconnu par le système CRISPR3/Cas. Par cette méthode, les auteurs ont démontré que les systèmes CRISPR/Cas et R/M agissent en synergie pour limiter les transferts horizontaux de gènes. Ainsi, l’absence de systèmes de défense fonctionnels (délétion des gènes codant la R/M ou Cas9), ou le partage du même système R/M entre la bactérie donneuse et la bactérie receveuse, permettent ces transferts horizontaux.
De plus, une limite du système R/M réside dans le fait qu’il peut être très rapidement débordé lors de l’entrée massive d’éléments génétiques étrangers dans la bactérie. En effet, une partie du matériel génétique entrant n’est pas dégradé par les endonucléases du système R/M et est au contraire méthylé de la même façon que le génome bactérien endogène. Cet élément génétique est alors protégé de toute dégradation et conservé par la bactérie comme étant du soi. Cet échappement au système R/M, confirmé dans cette étude, participe donc à la facilitation des transferts horizontaux de gènes en permettant l’acceptation d’éléments génétiques mobiles étrangers par un petit nombre de bactéries (débordement), puis leur transmission ultérieure lors des conjugaisons entre bactéries partageant le même système R/M.
Enfin, cette étude a également permis de démontrer que le segment CRISPR2 seul ne suffit pas à assurer une défense contre les transferts horizontaux de gènes, mais qu’il peut être fonctionnel en présence de l’endonucléase Cas9 associée au segment CRISPR1 (issue du système CRISPR1/Cas présent naturellement chez d’autres souches d’E. faecalis) du fait d’une origine évolutive commune.
Cette étude confirme bien que les transferts horizontaux de gènes sont facilités dans le cadre d’une déficience des systèmes de défense des bactéries. De ce fait, les bactéries « immunodéprimées » sont plus à même d’acquérir des multirésistances aux antibiotiques, ce qui est en accord avec les données génomiques des souches d’E. faecalis multirésistantes. Ces nouvelles connaissances pourraient permettre le développement de thérapies spécifiques à E. faecalis et ciblant les bactéries multirésistantes. Il est en effet possible d’envisager le recours à la phagothérapie, c’est-à-dire l’utilisation de bactériophages ciblant les souches pathogènes, comme démontré par Yosef et al. [ 8]. Le principe serait alors de profiter de l’absence, chez les souches pathogènes, de défense contre les transferts horizontaux de gènes pour permettre l’introduction, via la transduction virale, d’un système CRISPR/Cas fonctionnel dans ces bactéries et ciblant spécifiquement les gènes de résistances aux antibiotiques ou des gènes essentiels de leur génome. La phagothérapie se profile donc comme une stratégie possible contre les maladies nosocomiales provoquées par des bactéries pathogènes multirésistantes.