Les bactéries sont entourées d’une enveloppe cellulaire indispensable à leur protection, leurs échanges avec l’environnement, leur réponse comportementale, et au maintien de l’intégrité cellulaire. La structure et l’organisation de cette enveloppe varient en fonction du rang taxonomique des bactéries. Une majorité d’entre elles contient, dans son enveloppe, un polymère appelé peptidoglycane [1]. Ce polymère peut être épais chez les bactéries à Gram-positif ou beaucoup plus fin, et protégé par une seconde membrane externe, chez les bactéries à Gram-négatif (Figure 1). En fonction des différents phylums bactériens, la composition de l’enveloppe cellulaire comprend également une couche polysaccharidique extérieure, des protéines et des acides teichoïques et lipoteichoïques (bactéries à Gram positif) ou des lipopolysaccharides (bactéries à Gram négatif) [2].
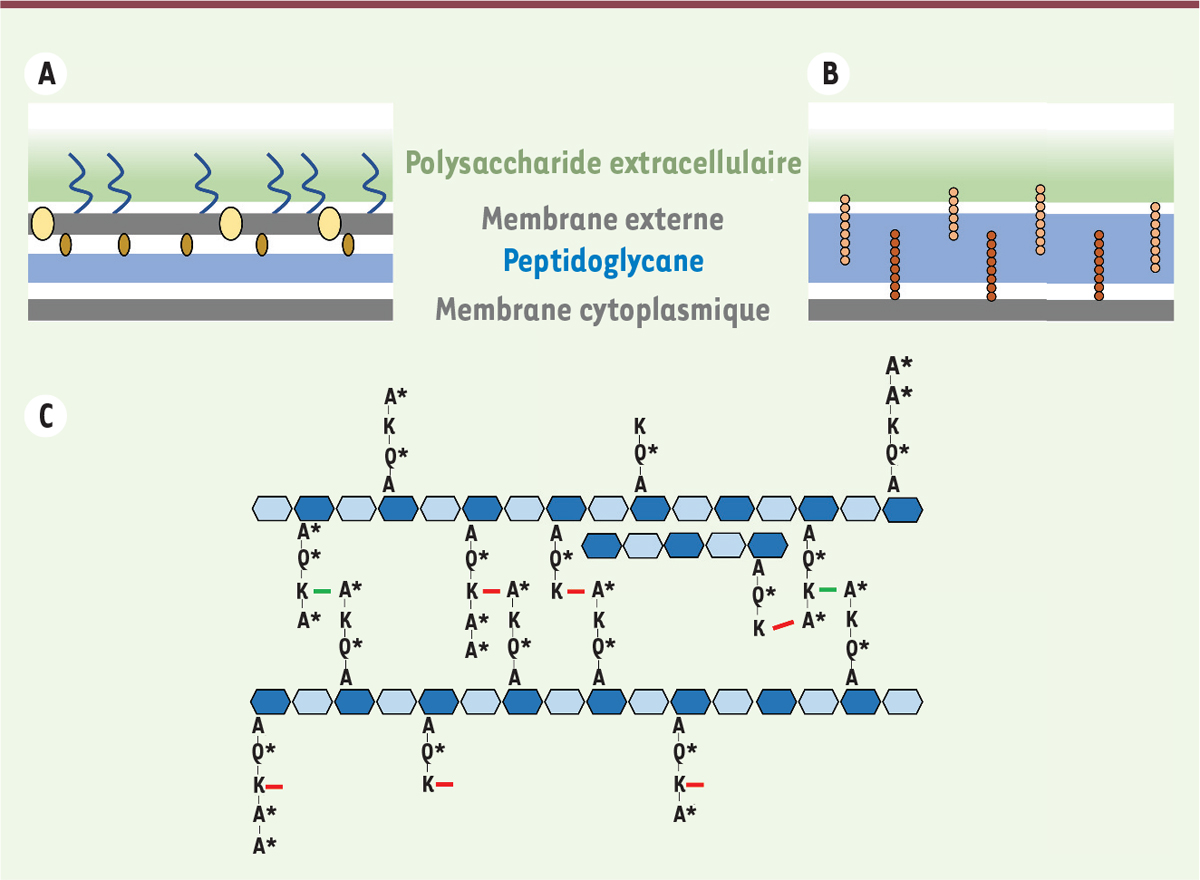
| Figure 1.
Structure et organisation de l’enveloppe cellulaire bactérienne. A. Bactéries à Gram négatif. Les ovales de couleur paille et marron symbolisent respectivement les protéines enchâssées dans la membrane externe, comme les porines, et la lipoprotéine de Braun, située à l’interface entre la face intérieure de la membrane externe et la couche de peptidoglycane. Les lipopolysaccharides ancrés dans la membrane externe sont représentés par des traits ondulés bleus. B. Bactéries à Gram positif. Les chaînes de perles de couleur saumon et brique symbolisent respectivement les acides téichoïques couplés à la couche de peptidoglycane et les acides lipotéichoïques ancrés dans la membrane plasmique. C. Composition et organisation de la couche de peptidoglycane. Les répétitions de N-acétyl glucosamine (hexagones bleu clair) et d’acide N-acétyl muramique (hexagones bleu foncé) composent les chaînes glucidiques. Ces dernières sont réticulées par des ponts peptidiques entre les peptides ancrés sur l’acide N-acétyl muramique de deux chaînes glucidiques différentes. La séquence des peptides est variable suivant les espèces bactériennes. La composition indiquée est celle retrouvée chez Streptococcus pneumoniae. A pour alanine, Q pour glutamine, K pour lysine. Les astérisques indiquent les acides aminés de configuration D. Les traits rouges symbolisent la réticulation directe entre la lysine en position 3 d’un peptide et l’alanine D en position 4 d’un autre peptide. Les traits verts symbolisent la réticulation indirecte via l’intermédiaire de ponts sérine/alanine ou alanine-alanine retrouvés chez le pneumocoque. |
Les propriétés biophysiques du peptidoglycane sont responsables de la forme cellulaire et assurent une protection mécanique nécessaire à l’intégrité de la cellule. Ce polymère est constitué de chaînes glucidiques, formées par la répétition de N-acétyl glucosamine et d’acide N-acétyl muramique, sur lesquelles se lient des chaînes peptidiques courtes composées d’acides aminés de configurations D et L [1]. Ces dernières forment des ponts peptidiques directs ou indirects, responsables de la réticulation des chaînes glucidiques (Figure 1). Ce maillage tridimensionnel constitue ainsi un réseau macromoléculaire souvent comparé à un véritable exosquelette qui entoure complètement la cellule bactérienne. Tout défaut dans l’assemblage de cette paroi est dommageable, voire fatal pour la cellule bactérienne. Ainsi, de nombreux antibiotiques actuels ciblent spécifiquement les enzymes impliquées dans la polymérisation et la réticulation du peptidoglycane.
L’assemblage du peptidoglycane nécessite des étapes de synthèse cytoplasmique assurant la formation d’un précurseur, appelé lipide II (Figure 2), qui est transloqué à l’extérieur de la cellule où il est intégré au polymère de peptidoglycane préexistant. Cette étape nécessite notamment l’action d’enzymes possédant des activités transglycosidases et/ou transpeptidases qui, respectivement, allongent et réticulent les chaînes glucidiques par des ponts peptidiques. Les enzymes responsables de ces étapes appartiennent à deux familles de protéines : les SEDS (shape, elongation, division and sporulation protein) et les PBP (penicillin-binding protein) (Figure 2) [3]. Les PBP représentent les plus anciennes cibles thérapeutiques majeures pour combattre les bactéries pathogènes. Elles sont, en effet, inhibées par des antibiotiques de type β-lactame, comme les pénicillines, les céphalosporines ou encore les carbapénèmes. La capacité de résistance des bactéries à ces antibiotiques est aujourd’hui souvent associée à des mutations des PBP [4]. Pour faire face aux infections dues aux bactéries résistantes, il est donc nécessaire d’imaginer de nouvelles stratégies d’inhibition des PBP permettant la déstabilisation et l’altération de l’assemblage du peptidoglycane.
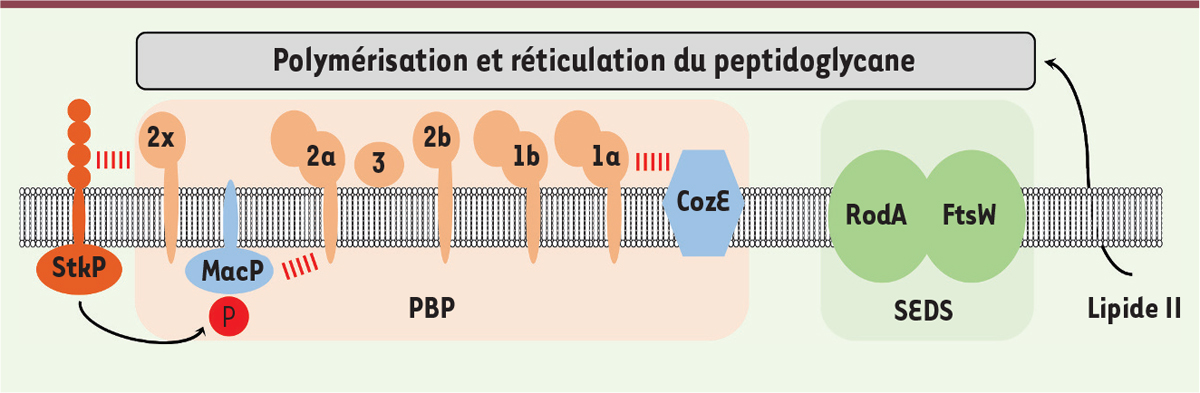
| Figure 2.
Assemblage et réticulation du peptidoglycane chez le pneumocoque. Le lipide II est synthétisé à la surface interne de la membrane plasmique. Après transport à travers la membrane, ce précurseur est pris en charges par les SEDS (shape, elongation, division and sporulation proteins en vert pâle) et les PBP (penicilin-binding protein, couleur saumon pâle) qui polymérisent et réticulent le polymère de peptidoglycane. Le pneumocoque possède deux SEDS (FtsW et RodA, ronds verts) et six PBP (transpeptidases 2x et 2b, transpeptidases/glycosyltransférases 1a, 1b et 2a, carboxypeptidase 3, ronds couleur saumon) nécessaires à cet assemblage. CozE, le régulateur négatif de PBP1a, et MacP (membrane-anchored cofactor of PBP2a), l’activateur de PBP2a, sont indiqués en bleu. Les traits rouges symbolisent les interactions entre les PBP et leur régulateur respectif. La serine/thréonine kinase StkP (marron) régule la fonction de MacP par phosphorylation et influence la fonction de PBP2x. |
Une perspective intéressante a émergé récemment à la suite de la découverte, chez les bactéries à Gram-négatif, des premiers activateurs de PBP, comme les lipo-protéines LpoA et LpoB, qui activent respectivement PBP1a et PBP1b d’Escherichia coli [5]. En ciblant ces activateurs par des molécules spécifiques, la fonction de la PBP associée pourrait ainsi être affectée, ce qui permettrait de lutter contre les bactéries résistantes aux antibiotiques actuels de type β-lactame. Les activateurs identifiés, comme LpoA et LpoB, semblent être conservés chez d’autres bactéries à Gram-négatif, mais ils ne sont pas retrouvés chez les bactéries à Gram-positif. Lors d’une étude récente réalisée en collaboration avec les équipes de D. Rudner et T. Bernhardt de l’université d’Harvard aux États-Unis, nous avons identifié une protéine membranaire, que nous avons nommée MacP (membrane-anchored cofactor of PBP2a), capable d’activer spécifiquement PBP2a, l’une des six PBP produites par la bactérie pathogène à Gram-positif Streptococcus pneumoniae (Figure 2) [6]. Cette bactérie, également appelée pneumocoque, est toujours considérée comme un pathogène de première importance, même en présence de traitements et de vaccins efficaces [7] (→).
(→) Voir la Synthèse de P. Léophonte et R. Garraffo, m/s hors série n° 3, août-septembre 2008, page 7
En 2017, l’Organisation mondiale de la santé a inclus cette bactérie dans la liste des 12 pathogènes bactériens les plus problématiques1. L’augmentation de la prépondérance des souches de pneumocoques résistantes à différents antibiotiques est également très inquiétante [8]. La caractérisation d’un activateur de l’activité d’une PBP, exprimée par le pneumocoque et d’autres bactéries à Gram-positif, pourrait permettre de concevoir de nouvelles molécules actives contre les bactéries résistantes aux antibiotiques.
Streptococcus pneumoniae produit trois PBP (PBP1a, PBP1b et PPBP2a) possédant simultanément des activités transpeptidase et transglycosidase, qui ne sont pas essentielles pour sa croissance. La délétion conjointe des gènes pbp1a et pbp2a est cependant létale pour la bactérie, indiquant que ces activités pourraient être fonctionnellement redondantes. Dans cette étude, la réalisation d’un crible génétique synthétique létal a permis de montrer que le gène macP est essentiel uniquement dans une souche dépourvue de pbp1a alors qu’il est facultatif dans une souche sauvage ou déficiente pour pbp2a [6]. De plus, la délétion de macP mime les effets de la délétion de pbp2a. En effet, en absence de macP ou de pbp2a, les cellules sont de plus petite taille et se lysent facilement. La spécificité de MacP envers PBP2a a été confirmée en mettant en évidence que la viabilité du mutant déficient pour macP n’est pas affectée par le niveau d’expression de pbp2a, mais est strictement dépendante de celui de pbp1a. L’utilisation d’une sonde fluorescente marquant spécifiquement les sites d’assemblage et de réticulation du peptidoglycane a également permis d’observer que la biogénèse de ce polymère est fortement impactée dans une souche sous-exprimant pbp1a et dépourvue de macP. L’analyse du réseau d’interaction de MacP a également démontré que MacP interagit directement et spécifiquement avec PBP2a. En effet, MacP n’est pas capable d’interagir avec d’autres facteurs membranaires, comme les protéines MreC, MreD et CozE2, nécessaires à la synthèse du peptidoglycane du pneumocoque. L’ensemble de ces observations indique donc que MacP est un activateur nécessaire et spécifique de la fonction de PBP2a (Figure 2).
La localisation cellulaire de MacP a ensuite été analysée pour en préciser sa fonction. Chez le pneumocoque, la synthèse de peptidoglycane est exclusivement localisée au centre de la cellule au niveau du septum de division. De manière cohérente, MacP et PBP2a présentent des localisations similaires, mais qui ne sont cependant pas affectées par leur délétion respective. Le recrutement de ces deux protéines au centre de la cellule fait donc appel à un mécanisme indépendant et encore inconnu. MacP promeut ainsi la fonction de PBP2a uniquement lorsque les deux protéines sont recrutées au septum de division. Il est intéressant de noter que MacP est phosphorylé par la sérine/thréonine-kinase-P (StkP) (Figure 2). Cette kinase est considérée comme le chef d’orchestre de la division cellulaire du pneumocoque en phosphorylant et en influençant la fonction de différentes protéines de la division cellulaire comme MapZ, DivIVA et EloR3 [9–11]. Le site unique de phosphorylation de MacP a été identifié et un mutant mimant un état permanent de déphosphorylation (mutation phospho-ablative) a été généré. Cette mutation bloque la croissance et entraîne la létalité d’une souche déficiente pour pbp1a. Néanmoins, MacP non-phosphorylable est toujours capable d’interagir avec PBP2a, indiquant que la phosphorylation de MacP n’est pas nécessaire à la formation du complexe PBP2a/MacP. Toutefois, cette phosphorylation pourrait influencer la fonction de PBP2a au sein du complexe d’assemblage et de réticulation du peptidoglycane.
Dans le futur, il faudra comprendre non seulement le mécanisme moléculaire d’activation de la fonction de PBP2a par MacP, mais également comment ce mécanisme est régulé par phosphorylation et coordonné à la fonction des autres protéines de division, elles-mêmes substrats de StkP. Il a été montré que cette kinase influence également, par interaction directe, la fonction de PBP2x, une PBP essentielle du pneumocoque [8]. Il a récemment été démontré que la protéine régulatrice CozE agit négativement sur PBP1a, dont la suractivité s’avère létale [12]. Dans ce contexte, on ne peut pas exclure que la fonction des trois autres PBP (PBP1b, PBP2b et PBP3) du pneumocoque puisse également dépendre de protéines régulatrices. Répondre à l’ensemble de ces questions permettra de comprendre les rouages de la division cellulaire et de la morphogenèse du pneumocoque et d’autres bactéries à Gram-positif. Cette connaissance fondamentale devrait ouvrir la voie à des travaux de recherche plus appliqués pour développer de nouvelles molécules altérant l’assemblage du peptidoglycane.