| |
| Med Sci (Paris). 34(8-9): 671–677. doi: 10.1051/medsci/20183408013.Mécanismes d’échappement des filovirus à l’immunité innée Baptiste Martin1 and Étienne Decroly1* 1Laboratoire architecture et fonction des macromolécules biologiques (AFMB) - UMR7257 CNRS, parc scientifique de Luminy, Aix-Marseille université, Marseille, France |
Vignette (Photo © Inserm - Peter Lenting). Les filovirus sont des virus à ARN négatif monosegmenté simple brin. Ils appartiennent à l’ordre des Mononegavirales qui comprend des pathogènes humains importants, comme le virus de la rage (RV), celui de la rougeole (MeV), des oreillons (MuV), ou le virus respiratoire syncytial (RSV), responsable de bronchiolites chez le nourrisson. La famille des Filoviridae est divisée en trois genres [1] : Ebolavirus, Marburgvirus et Cuevavirus. Au sein du genre Ebolavirus, cinq espèces virales ont été décrites, Zaire ebolavirus (EBOV), Sudan ebolavirus (SUDV), Tai Forest ebolavirus (TAFV), Bundibugyo ebolavirus (BDBV) et Reston ebolavirus (RESTV). Les genres Marburgvirus et Cuevavirus ne sont, chacun, constitués que d’une seule espèce, respectivement Marburg marburgvirus (MARV) et Lloviu cuevavirus (LLOV). Les virus RESTV et LLOV ne semblent pas pathogènes pour l’homme, contrairement aux autres espèces virales de la famille, qui ont été responsables d’épidémies de fièvres hémorragiques en Afrique. L’épidémie la plus importante a touché plusieurs pays d’Afrique de l’Ouest entre 2014 et 2016, faisant plus de 11 000 victimes, soulignant ainsi le manque d'outils thérapeutiques [2]. La sévérité des infections à filovirus a été largement documentée. Elle est notamment due à une réplication virale robuste et systémique associée à la subversion précoce de la réponse inflammatoire de l’hôte infecté. La réplication virale et la réponse antivirale massive et tardive se traduisent par l’apparition d’une forte fièvre et des fuites vasculaires. La réplication virale incontrôlée cache en réalité la capacité qu’a le virus d’inhiber la réponse immunitaire innée antivirale de l’hôte. L’objectif de cette revue est de présenter les bases moléculaires sur lesquelles reposent ces mécanismes de l’échappement des filovirus à l’immunité innée. Dans un premier temps, les voies de l’immunité innée seront brièvement présentées afin de dresser le portrait des premières lignes de défense de la cellule. Les bases moléculaires de différents mécanismes d’échappement récemment décrits chez les filovirus seront ensuite exposées. |
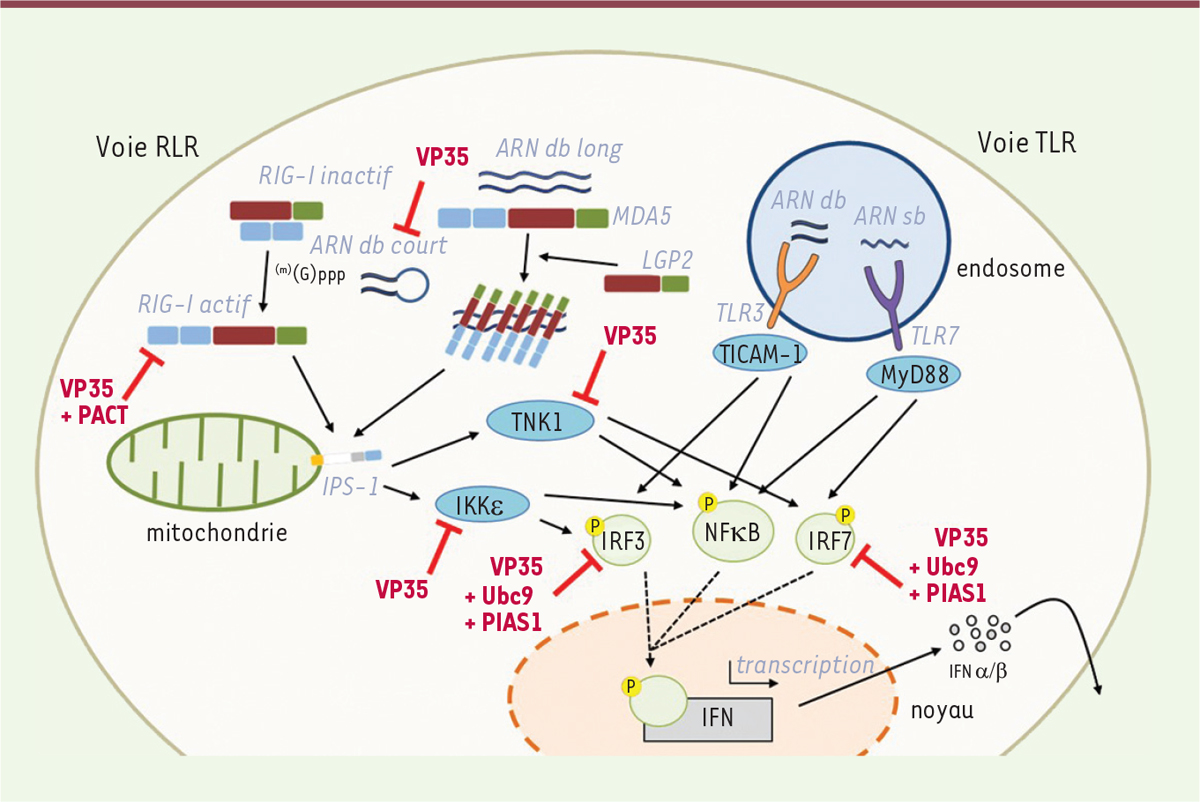
Bases moléculaires de l’immunité innée antivirale Les cellules eucaryotes ont acquis des mécanismes de défense limitant leur invasion par des pathogènes intracellulaires. Lorsqu’une cellule est infectée par un organisme exogène, comme un virus, sa première ligne de défense est constituée de mécanismes immunitaires précoces regroupés sous le nom d’immunité innée. La détection des pathogènes dépend de récepteurs de cette immunité innée (ou senseurs) à l’origine du déclenchement d’une cascade de signalisation permettant la synthèse d’effecteurs cellulaires antiviraux, et qui initient le recrutement des effecteurs de l’immunité dite adaptative, qui engendrera une réponse spécifique plus tardive, conférant une protection durable avec un effet mémoire contre l’agresseur. La détection des virus repose, entre autres, sur la reconnaissance de leurs acides nucléiques. Détection des acides nucléiques viraux L’immunité innée est activée en réponse à la reconnaissance de motifs moléculaires associés aux pathogènes (PAMP, pour pathogen-associated molecular patterns) par des récepteurs cellulaires appelés pattern recognition receptors (PRR). Deux classes principales de PRR sont impliquées dans la détection des ARN viraux (Figure 1) [ 3] : les RLR ( Rig-I-like receptors), des senseurs cytosoliques ubiquitaires de la famille du retinoic acid-inducible gene I (RIG-I), et des récepteurs membranaires de la famille des Toll-like receptors (TLR) exprimés plus spécifiquement dans les compartiments endosomaux ou à la surface des cellules immunitaires.
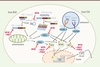 | Figure 1.
Détection des acides nucléiques exogènes et stimulation de la synthèse d’interférons. L’activation de l’immunité innée se fait pas la reconnaissance de motifs moléculaires exogènes. Deux voies sont responsables de la détection des virus via la reconnaissance de leurs acides nucléiques : la voie des RLR et la voie des TLR. La présence d’ARN double brin (db) court avec une extrémité 5’ triphosphate active RIG-I, tandis que l’ARN double brin long active MDA5. LGP2 participe à l’oligomérisation de MDA5 le long de son ARN cible. Ces deux RLR activent la molécule adaptatrice IPS-1 située à la surface des mitochondries qui active deux kinases, IKKε et TBK1. Ces kinases activent par phosphorylation les facteurs de transcription IRF3 et IRF7, respectivement, ainsi que NFκB. Ces facteurs migrent dans le noyau pour interagir avec le promoteur de nombreux gènes codant des facteurs antiviraux tels que les interférons. La présence d’ARN db ou simple brin stimule certains récepteurs endosomaux, TLR3 et TLR7 respectivement. TLR3 active TICAM-1 qui phosphoryle IRF3 et NFκB. TLR7 active MyD88 qui phosphoryle IRF7 et NFκB. Ces facteurs, une fois phosphorylés, migrent dans le noyau afin d’activer la transcription des IFN. Les filovirus codent la protéine VP35 qui est capable de bloquer différentes étapes de ces voies de signalisation en interagissant ou non avec d’autres partenaires cellulaires (PACT, protéines de SUMOylation Ubc9 et PIAS1) des protéines impliquées. RLR : RIG-I-like receptors ; TLR : Toll-like receptors ; db : double brin ; MDA5 : melanoma differentiation-associated gene 5 ; LGP2 : laboratory of genetics and physiology factor 2 ; IPS-1 : INF-α promotor stimulating factor 1 ; IKKε : IkB kinase ε ; TBK1 : TANK-binding kinase 1 ; IRF : interferon regulatory factor ; NFκB : nuclear factor κ-B ; TICAM-1 : Toll-like receptor adaptator molecule 1 ; MyD88 : myeloid differentiation primary response gene 88 ; IFN : interferon ; PACT : PKR activating protein. |
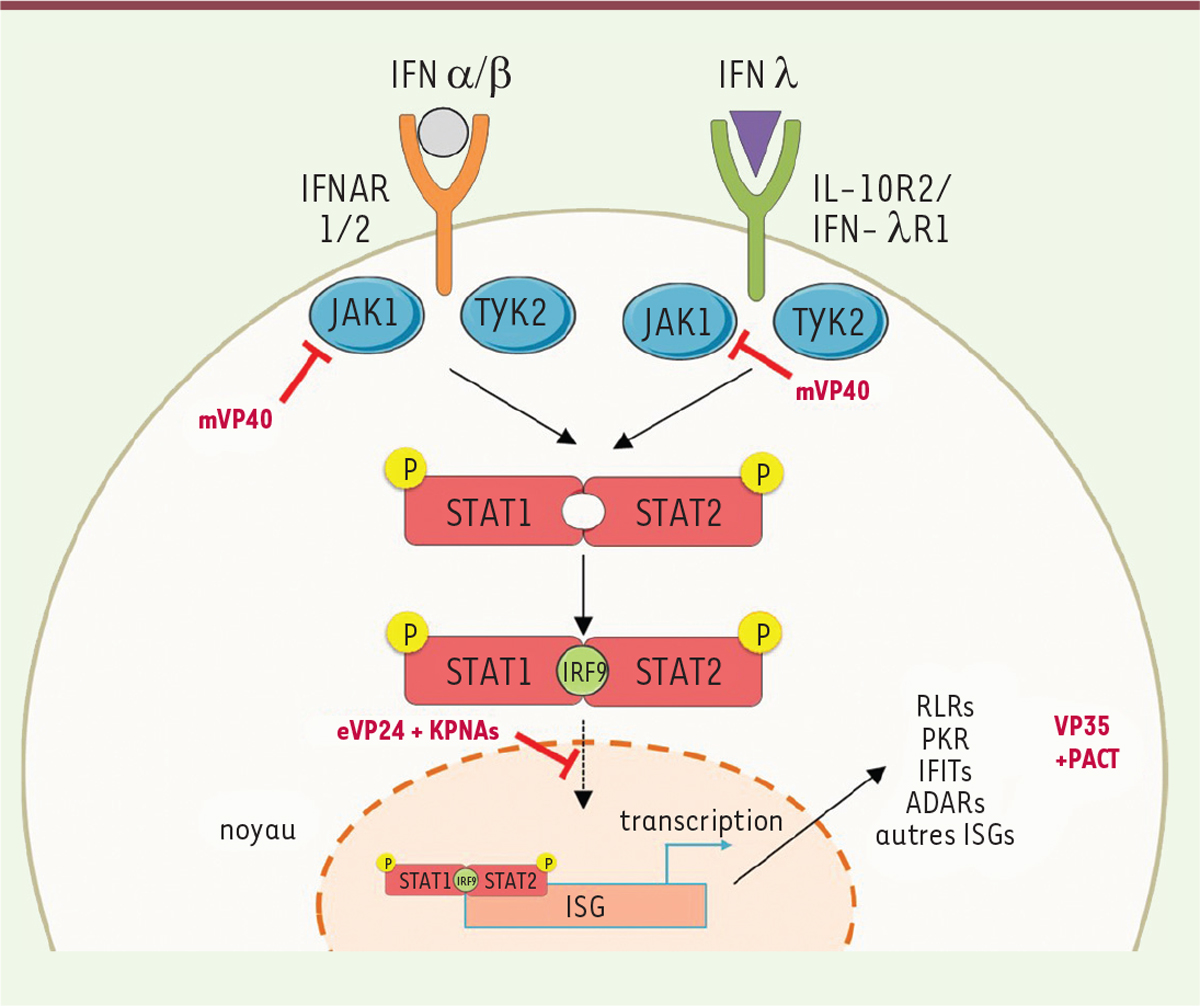
Le senseur RIG-I est une protéine cytosolique constituée de trois domaines distincts : une région N-terminale, composée d’un tandem de domaines destinés au recrutement et à l’activation de caspases (CARD, pour caspase recruitment domain), un domaine central portant une activité hélicase à ARN, avec un motif caractéristique, DEX(D/H), permettant de dérouler l’ARN en hydrolysant l’ATP grâce à une activité ATPase, et un domaine C-terminal dans lequel est intégré un domaine répresseur (RD). Lorsque RIG-I est inactivé, les domaines CARD sont séquestrés par le RD [4]. Quand RIG-I détecte un ARN exogène, le RD libère les domaines CARD qui sont alors ubiquitinylés, entraînant la tétramérisation du récepteur et l’induction de la cascade de signalisation à l’origine de la synthèse d’interférons (IFN) [5]. Récemment, il a été montré que RIG-I est capable de discriminer les ARN du « soi » du « non-soi » grâce à la détection des structures coiffe qui ne sont pas 2’ O-méthylées sur le premier nucléotide de l’ARN viral (contrairement aux ARN de l’hôte) [6]. MDA5 (melanoma differentiation-associated gene 5) est un autre senseur cytosolique qui possède une organisation en domaines similaire à celle de RIG-I. MDA5 ne contient par contre pas de domaine d’autorégulation : il adopterait une forme ouverte en l’absence de ligand et serait activé en présence de longs ARN double brin (db) [7]. MDA5 interagirait avec les ARN db sous la forme de monomère. Il s’oligomériserait ensuite en structure hélicoïdale [8], provoquant ainsi un changement de conformation exposant les CARD, qui initieraient le processus de signalisation induisant la production d’IFN de type I (IFN-α et IFN-β, en particulier). LGP2 (laboratory of genetics and physiology 2) diffère des deux autres senseurs car il ne possède pas de domaine CARD et ne transmet donc pas de signal de détection [7]. Son domaine hélicase lui permet cependant d’interagir avec des ARN exogènes, au milieu de la séquence (comme MDA5), et son domaine C-terminal de discriminer les différentes extrémités de l’ARN (comme RIG-I). LGP2 serait donc une molécule « chimère » entre RIG-I et MDA5. Au début de l’infection, LGP2 favoriserait la détection des ARN viraux par MDA5. Il contrôlerait ensuite négativement MDA5, à la suite de la stimulation des cellules par les IFN et de l’augmentation de son expression [9]. Les TLR sont constitués d’un domaine extracellulaire (ou luminal) contenant des régions riches en leucine (LRR) impliquées dans la reconnaissance des PAMP, un domaine transmembranaire et un domaine cytoplasmique de type Toll/interleukin-1 receptor (TIR) responsable de la transduction du signal conduisant à la production d’IFN [10]. Les TLR 3, 7, 8 et 9 sont localisés à la surface des cellules ou dans les endosomes. Ils permettent de détecter les acides nucléiques exogènes, dont ceux des virus. Le TLR 3 détecte l’ARN db par interactions électrostatiques non spécifiques entre les cycles imidazoles portés par 4 résidus histidines qu’il présente et les résidus phosphate de l’ARN. Les TLR 7 et 8 reconnaissent, quant à eux, les ARN viraux simple brin, riches en guanine et uracile (GU). Enfin, le TLR 9 décèle les motifs CpG (dinucléotides cytosine-guanine) non méthylés contenus dans l’ADN de certains virus. Contrairement aux RLR, la compartimentalisation des TLR dans l’endosome ou à la membrane plasmique fait de ces récepteurs non pas des senseurs discriminant le « soi » du « non soi », mais détectant des molécules qui ne devraient pas être présentes à la surface des cellules ou dans les endosomes. Activation de la synthèse d’interférons de type I L’activation des RLR par un ARN viral entraîne une cascade de signalisation permettant la production des IFN de type I (IFN-I ou α/β) (Figure 1) [ 11]. Les CARD de RIG-I et de MDA5 initient une cascade de signalisation via une interaction homotypique avec le domaine CARD du facteur de stimulation du promoteur des IFN-I (IPS-1, également appelé MAVS, VISA ou Cardif). L’activation des CARD d’IPS-1 entraîne son accumulation à la surface des mitochondries, ce qui active les kinases TBK1 ( TANK-binding kinase 1) et IKKε ( Iκ B kinase ε) qui, une fois activées, vont phosphoryler respectivement les facteurs de transcription IRF3 et IRF7 ( interferon regulatory factors 3 et 7) et le facteur nucléaire κ-B (NFκB). Ces facteurs migrent alors vers le noyau pour interagir avec les promoteurs des gènes cibles induisant la production des IFN-α et β et de cytokines pro-inflammatoires. Bien que les TLR utilisent des processus de détection différents, ils partagent avec les RLR les mécanismes de transduction du signal induisant la synthèse d’IFN (Figure 1) [12]. La détection de l’ARN viral par les TLR entraîne leur homodimérisation, permettant à leurs domaines TIR de recruter les adaptateurs cytosoliques nécessaires à la transduction du signal. Pour TLR 3, l’adaptateur est le facteur TICAM-1 (Toll-like receptor adaptator molecule 1) qui active les facteurs de transcription IRF3 et NFκB. Les TLR 7 et 9 recrutent MyD88 (myeloid differentiation primary response gene 88) qui induit l’activation de IRF7 et NFκB. Comme pour les RLR, ces facteurs intermédiaires sont à l’origine de la synthèse d’IFN-I et de cytokines pro-inflammatoires. Réponse à l’interféron On distingue trois types d’IFN selon leurs propriétés et leur origine [ 13]. Les IFN de type I (α, β, ω, κ et ε) sont produits par la plupart des cellules et ont une action antimicrobienne. Ils sont sécrétés à la suite de la détection d’antigènes dans la cellule. Ils agissent de manière autocrine et paracrine en s’associant au récepteur dimérique IFNAR1/2 ( interferon alpha and beta receptor subunit 1/2) (Figure 2) [ 13]. Cette interaction conduit au recrutement de JAK1 ( janus kinase 1) et de TYK2 ( tyrosine kinase 2) qui phosphorylent les facteurs de transcription STAT1 et STAT2 ( signal transducers and activators of transcription 1, 2). Ces deux protéines s’homo- ou s’hétérodimérisent, puis forment avec le facteur IRF9 un complexe hétéro-trimérique appelé ISGF3 ( interferon-stimulated gene factor 3), qui migre dans le noyau pour stimuler la transcription de différents ISG ( interferon-stimulated genes) parmi lesquels les gènes codant les RLR et les TLR, la PKR ( double-stranded RNA-dependent protein kinase), la 2’-5’-oligoadénylate synthétase (OAS), des déaminases spécifiques des adénosines d’ARN db (ADAR) ou encore d’autres interferon-induced proteins, comme les IFIT ( interferon-induced protein with tetratricopeptide repeats) qui ont un rôle dans l’inhibition précoce de la réplication du virus en séquestrant les ARN viraux. L’IFN de type II, ou IFN-γ, est, lui, principalement sécrété par certaines cellules immunitaires (monocytes, cellules NK [ natural killer], lymphocytes auxiliaires Th1 et lymphocytes cytotoxiques Tc). Il active et amplifie la réponse immunitaire cellulaire. Les IFN de type III, ou IFN-λ, ont été décrits plus récemment. Ils ont une action antivirale complémentaire à celle des IFN-I dans les tissus les plus exposés aux infections (tube digestif, système respiratoire, etc.).
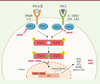 | Figure 2.
Voie de synthèse d’effecteurs en réponse à une stimulation par des interférons. La liaison des interférons (IFN) de type I (α ou β) à leur récepteur IFNAR1/2 permet le recrutement et l’activation des kinases JAK1 et TYK2 qui vont ensuite phosphoryler les protéines STAT1 et STAT2. L’activation des protéines STAT conduit à leur dimérisation puis au recrutement du facteur IRF9 qui forme alors un complexe hétérotrimérique qui est transféré au noyau pour stimuler la transcription d’ISGs. Cette voie de signalisation est également activée par les interférons de type 3 (IFN λ) via le récepteur IL10R2/IFN-λR1. Dans ce processus, les filovirus ont développé des mécanismes d’échappement. VP24 du virus Ebola (eVP24) bloque, par compétition avec l’agent de transfert, le transfert de STAT1/2-IRF9 au noyau. VP40 du virus Marburg (mVP40) bloque le recrutement et l’activation de la protéine JAK1. La protéine VP35 de l’ensemble des filovirus est également capable de bloquer l’effecteur PKR impliqué dans la dégradation des ARN. IFNAR : interferon-activated receptor ; JAK : Janus kinase ; TYK : tyrosine kinase ; STAT : signal transducer and activator of transcription ; IRF : interferon regulatory factor ; ISG : interferon-stimulated gene ; PKR : protein kinase R. |
|
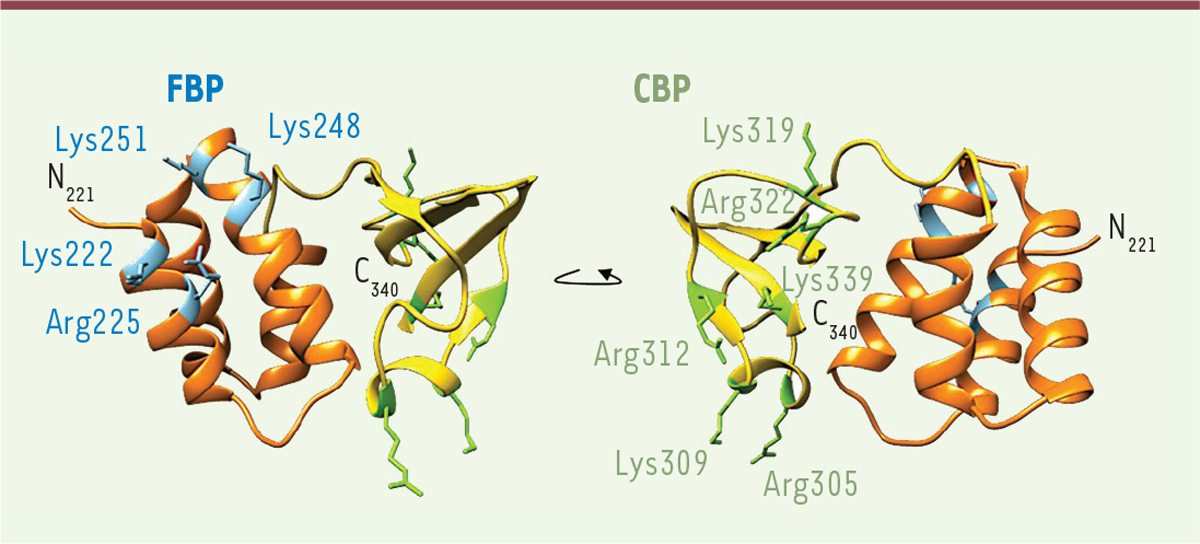
Subversion de la réponse immunitaire innée Les virus ont mis en place différentes stratégies originales afin de limiter leur détection par les senseurs de l’immunité innée et ainsi assurer leur réplication [14]. Chez les filovirus, plusieurs interférences avec la réponse antivirale ont été décrites récemment, impliquant diverses protéines virales et cellulaires (Figures 1 et 2). VP35, plaque tournante de la suppression de la signalisation cellulaire VP35 est un cofacteur de la polymérase L [ 15], qui joue également le rôle de facteur de virulence en inhibant l’immunité innée [ 16]. Chez l’ensemble des filovirus, VP35 est capable d’interférer avec les processus en amont et en aval de la détection des ARN par les RLR. VP35 est constitué de deux domaines [17, 18] : un domaine N-terminal permettant l’oligomérisation de la protéine, et un domaine C-terminal (CTD) appelé domaine d’inhibition des IFN (IID). La région comprise entre les résidus 220 et 340 du CTD a été définie comme la région minimale permettant le recrutement d’ARN db (Figure 3). Chez EBOV, le CTD est composé d’un sous-domaine en hélices α et d’un sous-domaine en feuillets β. L’étude des structures du CTD chez différents filovirus révèle une conservation structurale, avec la présence de régions riches en acides aminés basiques dans l’IID : le first basic patch (FBP) et le central basic patch (CBP). Ces régions sont impliquées dans la reconnaissance d’ARN db et dans l’inhibition des voies d’activation des IFN. Le FBP est cependant conservé uniquement chez les virus du genre Ebolavirus et semble plutôt impliqué dans la réplication virale [19]. Malgré la conservation globale de l’IID de VP35 chez les filovirus, quelques différences structurales et biochimiques ont été décrites qui semblent responsables d’une différence d’affinité avec les ARN db [17]. VP35 d’EBOV pourrait en effet se lier à la fois aux extrémités et à la séquence d’ARN db. VP35 de MARV ne reconnaîtrait, lui, que la séquence d’ARN db sans se lier à ses extrémités [20]. VP35 d’EBOV pourrait donc masquer les extrémités des ARN db et les rendre inaccessibles aux senseurs cytosoliques RLR.
 | Figure 3.
Structure du domaine IID de la protéine virale VP35 d’EBOV. La protéine VP35 des filovirus est constituée de deux domaines : un domaine N-terminal (1-220), impliqué dans l’oligomérisation de VP35 et la réplication virale, et un domaine C-terminal (221-340), responsable de l’inhibition de la réponse interféron (IID). La structure de ce dernier a été résolue par cristallographie aux rayons X (PDB : 3FKE). Elle révèle une organisation en un sous-domaine N-terminal en hélices α (orange) et un sous-domaine C-terminal en feuillets β (jaune). Cette structure est conservée chez les filovirus, notamment une région centrale riche en acides aminés basiques (central basic patch, ou CBP) (vert) ainsi qu’une seconde région, conservée uniquement chez les virus du genre Ebolavirus, en N-terminal (first basic patch, ou FBP) (bleu). Ces régions interviennent dans la reconnaissance d’ARN double brin et dans l’inhibition des voies d’activation des interférons. |
VP35 peut également interagir avec PACT (PKR-activating protein) (Figure 1 et 2), une protéine multifonctionnelle impliquée dans la régulation de RIG-I et de la PKR (ds-RNA-dependent protein kinase). PKR est codée par un ISG et porte une activité kinase dépendante des ARN db [21]. In vitro, VP35 bloque l’interaction entre PACT, RIG-I et PKR, empêchant ainsi l’activation de ces protéines cellulaires. Cette inhibition fait intervenir la même région basique permettant l’interaction de VP35 avec les ARN db, suggérant un mécanisme de régulation dépendant des concentrations de VP35, de PACT et des ARN db. VP35 interagit également avec les kinases IKKε et TBK1, comme un « substrat-leurre », entraînant sa phosphorylation en limitant ainsi l’activation d’IRF3 (Figure 1) [22]. VP35 interagit aussi avec IRF3 ou IRF7, l’enzyme E2 Ubc9 SUMO-conjuguée, et la ligase SUMO-E3 PIAS1, favorisant la SUMOylation des IRF (Figure 1) [23]. Cette modification altère la cascade de signalisation en aval des IRF et donc la synthèse d’IFN. Le domaine IID de VP35 joue donc un rôle clé pour les filovirus puisqu’en inhibant différents acteurs de la détection des ARN viraux par les cellules de l’hôte infecté, il permet à VP35 d’assurer ses différentes fonctions dans le cycle réplicatif des virus. L’étude structurale d’IID apparaît donc être un point clé dans le développement d’antiviraux. Dérégulation de la réponse à l’IFN En plus de la prévention de l’activation de l’immunité innée par VP35, les filovirus peuvent également bloquer la réponse des cellules aux IFN, induisant alors une forte répression de la synthèse des effecteurs antiviraux cellulaires. Différentes protéines virales interfèrent en effet avec la voie JAK/STAT, fondamentale pour la transduction du signal en réponse aux IFN. Chez les virus du genre Ebolavirus, VP24 (eVP24), impliquée dans les événements précoces d’assemblage du virus [24], participe également à l’inhibition de la réponse aux IFN [25, 26] (Figure 2). eVP24 peut en effet interagir avec KPNA 1, 5 et 6, trois protéines de la famille des karyophérines α (KPNA) [27]. Ces protéines se lient à STAT1 afin de transporter vers le noyau cellulaire le complexe activé STAT1/2-IRF9. Or, eVP24 se lie sur le même site d’interaction des KPNA que STAT1, ce qui, par compétition, bloque l’importation du complexe activé et est à l’origine de l’inhibition de la synthèse des ISG. Contrairement à eVP24, VP24 de MARV ne semble pas interagir avec les KPNA ni affecter l’expression d’ISG [28]. Cependant, chez ce virus, une autre protéine, VP40 (mVP40), bloque spécifiquement l’activation de JAK1 (Figure 2) [28]. Le mécanisme d’interférence de VP40 avec JAK1 n’est pas encore élucidé, et aucune interaction directe entre mVP40 et JAK1 n’a été démontrée. Ainsi, contrairement à EBOV, MARV empêche la transduction du signal induisant la synthèse des ISG par inhibition de la phosphorylation de STAT1 et STAT2 et non du transfert du complexe activé dans le noyau. Les filovirus ont également développé des mécanismes de défense contre les ISG produites en réponse aux IFN. À ce jour, deux cibles ont été identifiées : la PKR [29], dont nous avons présenté le mécanisme d’inhibition par VP35, et la téthérine, un facteur de restriction cellulaire qui bloque l’assemblage et le bourgeonnement de certains virus, empêchant la libération des particules virales [30]. En présence de VP40, la libération des particules virales infectieuses révèle un échappement à l'action de la téthérine, vraisemblablement grâce à la glycoprotéine virale de surface GP. Les mécanismes moléculaires permettant à la GP d’interférer avec la téthérine sont encore mal documentés, mais la GP ne semble influer ni sur son expression, ni sur son association au sein de radeaux lipidiques cellulaires [31]. |
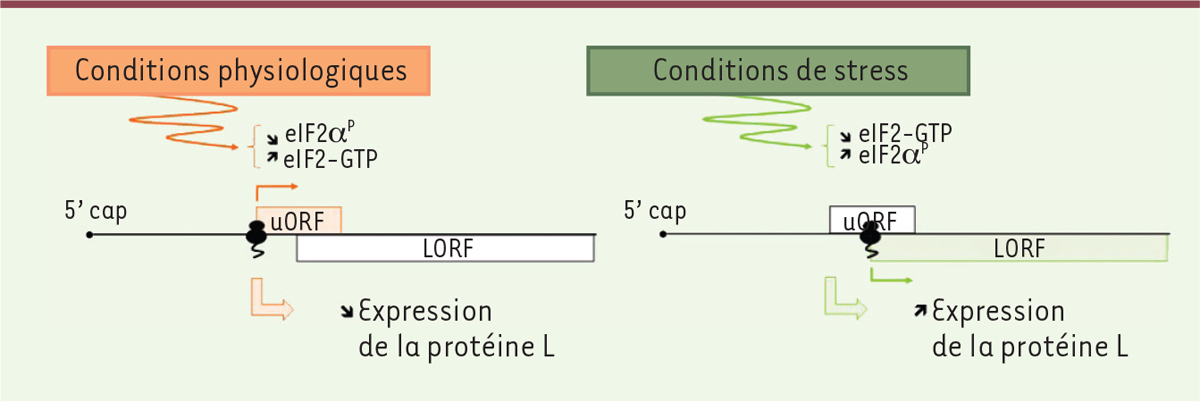
Autres mécanismes subversifs des réponses cellulaires à l’infection L’efficacité de réplication des filovirus ne résulte pas uniquement de leur capacité à inhiber les mécanismes de l’immunité innée. Des études récentes ont en effet révélé que ces virus contrôlent également la machinerie de traduction cellulaire (Figure 4). Les filovirus produisent des ARN messagers (ARNm) qui possédent de longues régions non traduites à leurs extrémités 5’ et 3’ (5’UTR et 3’UTR). Les 5’UTR des ARNm de VP35, VP30, VP24 et la protéine L d’EBOV contiennent un motif AUG (uAUG) qui marque le début d’une petite phase ouverte de lecture (uORF) [32]. L’expression de ces uORF semble réprimer la traduction des ORF codant les protéines virales, comme cela a été décrit pour certaines protéines cellulaires [33]. Un rôle similaire a ainsi été proposé pour les uORF mises en évidence chez les filovirus. En système réplicon, la traduction de la protéine virale L a été observée dans des conditions de stress où le facteur d’initiation eIF-2α (eukaryotic initiation factor 2) est phosphorylé, et la mutation de son uORF conduit à une diminution significative de la réplication d’EBOV. Bien que cela n’ait pas été démontré, il a été proposé que le virus pourrait augmenter la survie des cellules infectées par ce mécanisme [34].
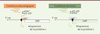 | Figure 4.
Subversion de la machinerie de traduction cellulaire pour assurer la réplication des filovirus. Plusieurs ARNm du virus Ebola possèdent de courtes phases ouvertes de lecture (uORF) en amont de la séquence codante pour les différentes protéines virales. Dans des conditions physiologiques, l’uORF présent dans l’ARNm codant la protéine L atténue sa traduction et ce, du fait d’une initiation efficace au codon AUG de l’uORF. En conditions de stress, lors d’une activation de la réponse immunitaire innée par exemple, le taux de phosphorylation du facteur eIF2α (eIF2α-P) augmente, ce qui diminue l’efficacité de l’initiation pour l’uORF. Le ribosome est alors capable d’initier la traduction de l’ORF de la protéine L. |
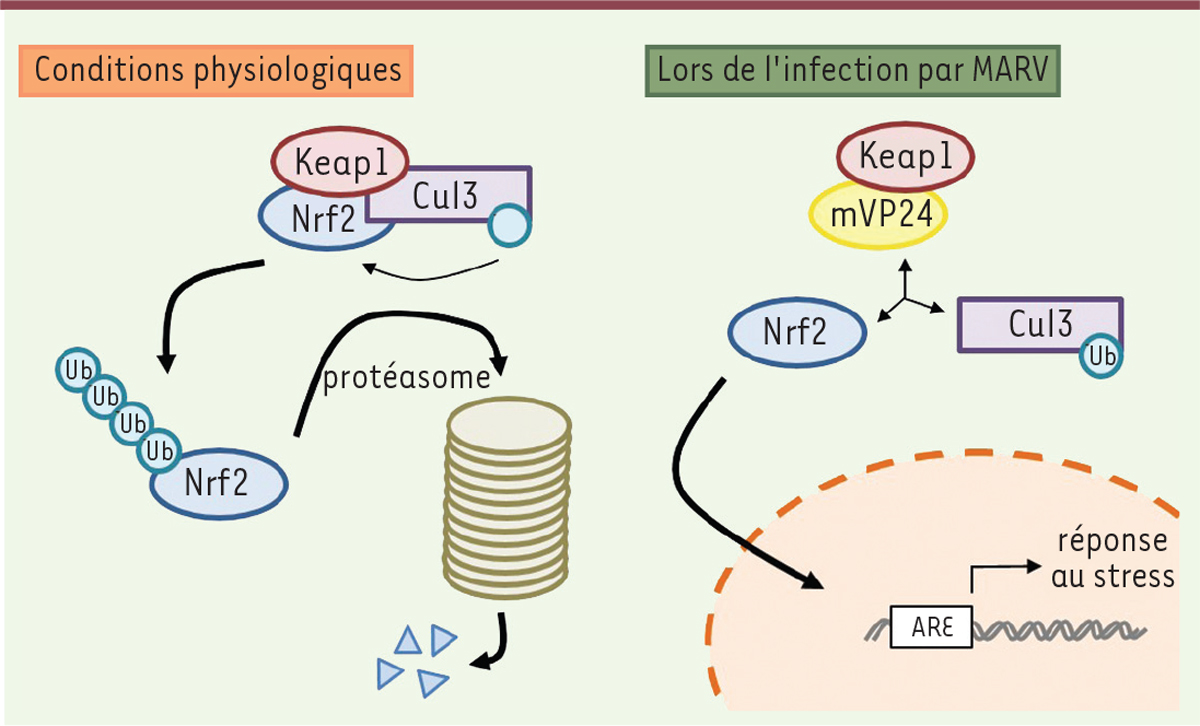
Un autre exemple de mécanisme de subversion des processus cellulaires repose sur l’interaction de mVP24 avec la protéine cellulaire Keap1 (Kelch-like ECH-associated protein 1) entraînant sa modulation (Figure 5) [34,35]. Keap1 oriente l’ubiquitine ligase Cul3 (Cullin 3) vers différentes protéines, comme Nrf2 (nuclear factor [erythroid-derived 2]-like 2), un facteur de transcription responsable de l’activation de gènes impliqués dans la réponse anti-oxydante et dont le promoteur présente un antioxidant response element (ARE). Les gènes sous contrôle de ce type de promoteur permettent aux cellules de récupérer après un stress. En conditions physiologiques, Nrf2 est continuellement dégradée par son ubiquitination via Keap1 et Cul3. Or, en situation de stress, l’interaction entre Nrf2 et Keap1/Cul3 est perdue, entraînant la transcription des gènes dont les promoteurs présentent un ARE [36]. mVP24 interagit avec Keap1 sur le même site d’interaction que Nrf2, induisant ainsi l’expression des gènes de stress [34]. L’intérêt de ce mécanisme dans la survie du virus n’a pas été démontré. Il est proposé qu’il augmenterait la survie des cellules infectées. Keap1 induit également l’ubiquitinylation de la protéine kinase IKKβ (IκB kinase β) qui, comme Nrf2, est inhibée par mVP24 [37]. Cette inhibition entraîne l’activation de la voie NFκB, dont l’implication dans la pathogenèse de MARV reste cependant à élucider.
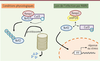 | Figure 5.
Implication de mVP24 dans la subversion du stress cellulaire. Keapl recrute l’ubiquitine ligase Cul3 et le facteur de transcription Nrf2, responsable de l’activation des promoteurs contenant un antioxidant response element (ARE). En conditions physiologiques, Nrf2 est continuellement dégradé par le protéasome du fait de son ubiquitination par Keap1/Cul3. Or, en situation de stress, l’interaction Nrf2-Keap1/Cul3 est perdue, entraînant la transcription des gènes sous contrôle d’un promoteur ARE. Ces gènes permettent alors aux cellules de récupérer à la suite d’un stress. mVP24 mime cet état de stress en interagissant avec Keap1 sur le même site d’interaction que Nrf2, ce qui entraîne l’expression des gènes de réponse au stress. Keap1 : Kelch-like Ech-associated protein 1 ; Cul3 : Cullin 3. |
|
Il est donc clair que les filovirus ont développé, grâce à leurs protéines, de nombreuses stratégies leur permettant de se prémunir de leur détection par l’immunité innée et d’adapter leurs mécanismes de réplication à leur hôte. Ces mécanismes de protection touchent tous les niveaux de la cascade de signalisation menant à la synthèse d’IFN puis d’ISG. Les ARN viraux portent également des modifications, comme la coiffe des ARNm, qui sont impliquées dans la discrimination du « soi » et du « non-soi », ce qui offre également d’autres perspectives pour le développement de stratégies antivirales. |
Les auteurs déclarent n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
|
1. Kuhn JH. Guide to the correct use of filoviral nomenclature . Curr Top Microbiol Immunol. 2017; ; 411 : :447.–460. 2. Baize S, Pannetier D, Oestereich L, et al. Emergence of Zaire Ebola virus disease in Guinea . N Engl J Med. 2014; ; 371 : :1418.–1425. 3. Schlee M, Hartmann G. Discriminating self from non-self in nucleic acid sensing . Nat Rev Immunol. 2016; ; 16 : :566.–580. 4. Kowalinski E, Lunardi T, McCarthy AA, et al. Structural basis for the activation of innate immune pattern-recognition receptor RIG-I by viral RNA . Cell. 2011; ; 147 : :423.–435. 5. Jiang X, Kinch LN, Brautigam CA, et al. Ubiquitin-induced oligomerization of the RNA sensors RIG-I and MDA5 activates antiviral innate immune response . Immunity. 2012; ; 36 : :959.–973. 6. Devarkar SC, Wang C, Miller MT, et al. Structural basis for m7G recognition and 2’-O-methyl discrimination in capped RNAs by the innate immune receptor RIG-I . Proc Natl Acad Sci USA. 2016; ; 113 : :596.–601. 7. Uchikawa E, Lethier M, Malet H, et al. Structural analysis of dsRNA binding to anti-viral pattern recognition receptors LGP2 and MDA5 . Mol Cell. 2016; ; 62 : :586.–602. 8. Wu B, Peisley A, Richards C, et al. Structural basis for dsRNA recognition, filament formation, and antiviral signal activation by MDA5 . Cell. 2013; ; 152 : :276.–289. 9. Rodriguez KR, Bruns AM, Horvath CM. MDA5 and LGP2 : accomplices and antagonists of antiviral signal transduction . J Virol. 2014; ; 88 : :8194.–8200. 10. Zhang Z, Ohto U, Shimizu T. Toward a structural understanding of nucleic acid-sensing Toll-like receptors in the innate immune system . FEBS Lett. 2017; ; 591 : :3167.–3181. 11. Fensterl V, Chattopadhyay S, Sen GC. No love lost between viruses and interferons . Annu Rev Virol. 2015; ; 2 : :549.–572. 12. Satoh T, Akira S. Toll-like receptor signaling and its inducible proteins . Microbiol Spectr. 2016 ; :4.. 13. Wang BX, Fish EN. The yin and yang of viruses and interferons . Trends Immunol. 2012; ; 33 : :190.–197. 14. García-Sastre A. Ten strategies of interferon evasion by viruses . Cell Host Microbe. 2017; ; 22 : :176.–184. 15. Martin B, Canard B, Decroly E. Filovirus proteins for antiviral drug discovery : structure/function bases of the replication cycle . Antiviral Res. 2017; ; 141 : :48.–61. 16. Leung DW, Prins KC, Basler CF, Amarasinghe GK. Ebolavirus VP35 is a multifunctional virulence factor . Virulence. 2010; ; 1 : :526.–531. 17. Ramanan P, Edwards MR, Shabman RS, et al. Structural basis for Marburg virus VP35-mediated immune evasion mechanisms . Proc Natl Acad Sci USA. 2012; ; 109 : :20661.–20666. 18. Edwards MR, Liu G, Mire CE, et al. Differential regulation of interferon responses by Ebola and Marburg virus VP35 proteins . Cell Rep. 2016; ; 14 : :1632.–1640. 19. Prins KC, Binning JM, Shabman RS, et al. Basic residues within the ebolavirus VP35 protein are required for its viral polymerase cofactor function . J Virol. 2010; ; 84 : :10581.–91. 20. Messaoudi I, Amarasinghe GK, Basler CF. Filovirus pathogenesis and immune evasion : insights from Ebola virus and Marburg virus . Nat Rev Microbiol. 2015; ; 13 : :663.–676. 21. Kok KH, Lui PY, Ng MH, et al. The double-stranded RNA-binding protein PACT functions as a cellular activator of RIG-I to facilitate innate antiviral response . Cell Host Microbe. 2011; ; 9 : :299.–309. 22. Prins KC, Cárdenas WB, Basler CF. Ebola virus protein VP35 impairs the function of interferon regulatory factor-activating kinases IKKepsilon and TBK-1 . J Virol. 2009; ; 83 : :3069.–3077. 23. Chang TH, Kubota T, Matsuoka M, et al. Ebola Zaire virus blocks type I interferon production by exploiting the host SUMO modification machinery . PLoS Pathog. 2009; ; 5 : :e1000493.. 24. Martin B, Reynard O, Volchkov V, Decroly E. Filovirus proteins for antiviral drug discovery : Structure/function of proteins involved in assembly and budding . Antiviral Res. 2018; ; 150 : :183.–192. 25. Reid SP, Leung LW, Hartman AL, et al. Ebola virus VP24 binds karyopherin alpha1 and blocks STAT1 nuclear accumulation . J Virol. 2006; ; 80 : :5156.–5167. 26. Reid SP, Valmas C, Martinez O, et al. Ebola virus VP24 proteins inhibit the interaction of NPI-1 subfamily karyopherin alpha proteins with activated STAT1 . J Virol. 2007; ; 81 : :13469.–13477. 27. Xu W, Edwards MR, Borek DM, et al. Ebola virus VP24 targets a unique NLS binding site on karyopherin alpha 5 to selectively compete with nuclear import of phosphorylated STAT1 . Cell Host Microbe. 2014; ; 16 : :187.–200. 28. Valmas C, Grosch MN, Schümann M, et al. Marburg virus evades interferon responses by a mechanism distinct from ebola virus . PLoS Pathog. 2010; ; 6 : :e1000721.. 29. Feng Z, Cerveny M, Yan Z, He B. The VP35 protein of Ebola virus inhibits the antiviral effect mediated by double-stranded RNA-dependent protein kinase PKR . J Virol. 2007; ; 81 : :182.–192. 30. Kühl A, Banning C, Marzi A, et al. The Ebola virus glycoprotein and HIV-1 Vpu employ different strategies to counteract the antiviral factor tetherin . J Infect Dis. 2011; ; 204 ((suppl 3)) : :S850.–s860. 31. Lopez LA, Yang SJ, Exline CM, et al. Anti-tetherin activities of HIV-1 Vpu and Ebola virus glycoprotein do not involve removal of tetherin from lipid rafts . J Virol. 2012; ; 86 : :5467.–5480. 32. Shabman RS, Hoenen T, Groseth A, et al. An upstream open reading frame modulates ebola virus polymerase translation and virus replication . PLoS Pathog. 2013; ; 9 : :e1003147.. 33. Wek RC, Jiang HY, Anthony TG. Coping with stress : eIF2 kinases and translational control . Biochem Soc Trans. 2006; ; 34 : :7.–11. 34. Page A, Volchkova VA, Reid SP, et al. Marburgvirus hijacks nrf2-dependent pathway by targeting nrf2-negative regulator keap1 . Cell Rep. 2014; ; 6 : :1026.–1036. 35. Edwards MR, Johnson B, Mire CE, et al. The Marburg virus VP24 protein interacts with Keap1 to activate the cytoprotective antioxidant response pathway . Cell Rep. 2014; ; 6 : :1017.–1025. 36. Copple IM. The Keap1-Nrf2 cell defense pathway : a promising therapeutic target? Adv Pharmacol. 2012; ; 63 : :43.–79. 37. Edwards MR, Basler CF. Marburg Virus VP24 protein relieves suppression of the NF-κB pathway through interaction with Kelch-like ECH-Associated Protein 1 . J Infect Dis. 2015; ; 212 ((suppl 2)) : :S154.–S159. |