L’infection par le virus Ébola conduit à des fièvres hémorragiques souvent fatales. Depuis sa découverte en 1976 en République Démocratique du Congo, plus d’une vingtaine d’épidémies d’Ébola ont été recensées en Afrique. La plus importante a eu lieu en 2015-2016, entraînant le décès de plus de 11 000 personnes [1]. En raison du risque de réémergence de ce virus, et en l’absence de vaccins et d’agents thérapeutiques approuvés, la recherche de médicaments ciblant le virus Ébola est une priorité pour l’Organisation mondiale de la santé. Le virus Ébola est un virus enveloppé possédant un ARN génomique simple brin de polarité négative et appartient à l’ordre les Mononegavirales. Cet ordre est divisé en 5 familles de virus contenant des pathogènes humains importants, comme les virus de la rage, de la rougeole, ou encore le virus respiratoire syncytial (VRS). Le virus Ébola fait partie de la famille des Filovirus (→) [2] et appartient au genre Ebolavirus qui contient 5 espèces : Zaïre, Soudan, Taï Forest, Bundibugyo et Reston.
(→) Voir la Synthèse de B. Martin et E. Decroly, m/s n° 8-9, août-septembre 2018 page 671
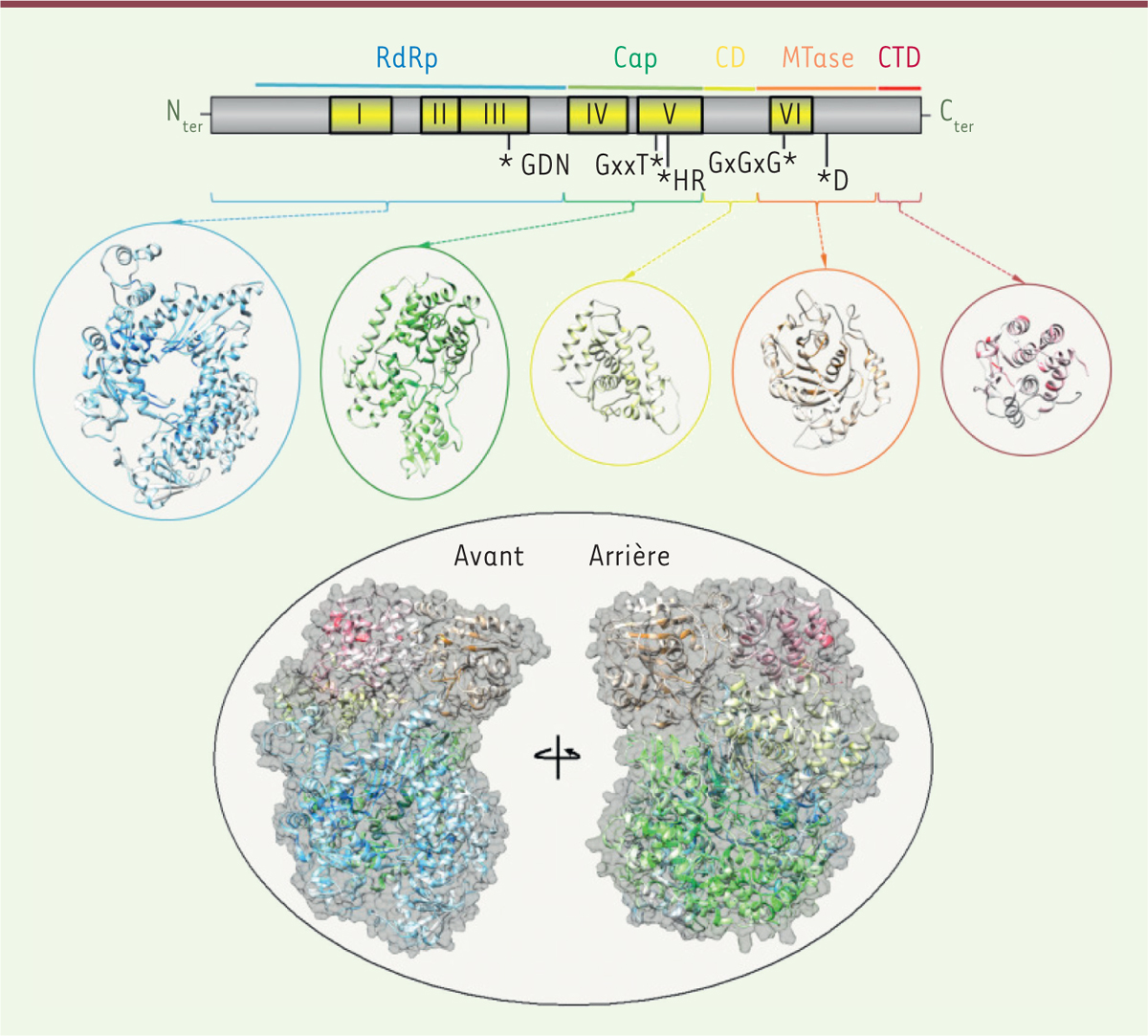
Le génome des filovirus code sept protéines virales produites à partir d’ARN messagers (ARNm) transcrits de manière séquentielle. Parmi ces protéines, la protéine « Large » (L) est un des acteurs majeurs de la propagation de l’infection puisqu’elle contient les activités enzymatiques nécessaires à la réplication/transcription et à la maturation des ARN viraux [3]. N’ayant pas d’homologues cellulaires, la protéine L constitue une cible antivirale attractive. Cette protéine contient six régions conservées (CR) entrecoupées de régions variables. La structure, récemment résolue par cryo-microscopie, de la protéine L du virus VSV (virus de la stomatite vésiculaire) indique que cette protéine est organisée en 4 domaines structuraux distincts (Figure 1) [4]. L’extrémité N-terminale de la protéine (régions CRI à CRIII) forme le domaine ARN polymérase ARN-dépendante (RdRp) intimement intriqué avec le domaine Cap (régions CRIV et CRV) qui porte l’activité polyribonucléotidyltransférase (PRNTase) nécessaire à la synthèse de la structure coiffe à l’extrémité 5’ des ARNm. L’extrémité C-terminale de la protéine L contient successivement les domaines connecteur (CD), méthyltransférase (MTase) (CRVI) et C-terminal (CTD). La coiffe synthétisée par les mononégavirus est donc constituée d’une molécule de guanosine méthylée en position N7 reliée à l’extrémité 5’ des ARNm par une liaison 5’-5’ triphosphate. Cette coiffe est également méthylée en position 2’O du ribose du premier nucléotide [5]. Par analogie avec d’autres Mononegavirales, le domaine MTase des filovirus a été proposé comme participant au processus de méthylation de la structure coiffe. Ce domaine contient un motif de reconnaissance d’une molécule donneuse de méthyle (S-adénosyl méthionine, SAM) à savoir Gly-X-Gly-X-Gly (X étant un acide aminé quelconque) et une tétrade catalytique caractéristique des 2’O-MTases (Lys-Asp-Lys-Glu). La présence de cette structure coiffe à l’extrémité des ARNm viraux est essentielle pour la propagation de l’infection virale. En effet, elle protège les ARNm viraux de la dégradation par des 5’-exonucléases cellulaires et assure le recrutement du facteur d’initiation de la traduction eIF4E, nécessaire à la traduction des ARNm en protéines [5]. Par ailleurs, il a été démontré que la 2’O-méthylation de cette structure coiffe était un marqueur du « soi » jouant un rôle dans le contrôle de la réponse antivirale [6]. Des études récentes ont démontré que la 2’O-méthylation des structures coiffes empêche la détection des ARN viraux par les senseurs cytoplasmiques de l’immunité innée tels que les protéines RIG-I (retinoic-acid-inducible protein 1) et/ou MDA5 (melanoma-differentiation-associated gene 5) et limite ainsi la production d’interféron [2]. De plus, il a été montré qu’il existe, parmi les ISG (interferon-stimulated gene), des facteurs de restriction comme les protéines IFIT (IFN-induced proteins with tetratricopeptide repeats), qui reconnaissent et séquestrent les ARN possédant des structures coiffées incomplètement méthylées [7]. Les enzymes virales impliquées dans la synthèse des structures coiffes et leurs méthylations constituent donc des cibles thérapeutiques pertinentes. En effet, l’inhibition de ces enzymes devrait d’une part bloquer la réplication virale et, d’autre part, induire une détection précoce des agents infectieux par les mécanismes de l’immunité innée induisant une réponse antivirale protectrice.
| Figure 1. Domaines fonctionnels de la protéine L des Mononegavirales. Les régions conservées ont été identifiées après alignements multiples par bioinformatique. La structure présentée est celle de la protéine du virus VSV (virus de la stomatite vésiculaire) résolue par cryo-microscopie [3] qui révèle la présence de 5 domaines structuraux : le domaine ARN polymérase ARN-dépendante (RdRp) intimement intriqué avec le domaine Cap (régions CRIV et CRV) qui porte l’activité polyribonucléotidyltransférase (PRNTase) ; l’extrémité C-terminale de la protéine L contient successivement les domaines connecteur (CD), méthyltransférase (MTase) contenant la région CRVI et C-terminale (CTD). |
La structure et l’activité MTase de la coiffe ont été récemment caractérisées chez le virus hMPV (human metapneumovirus, un autre virus de l’ordre des Mononegavirales) [8], mais nous manquions d’informations structurales et fonctionnelles concernant les activités MTases du virus Ébola. Nous avons donc cloné le domaine C-terminal de la protéine L du virus Ébola (MTase et CTD) pour le produire en système bactérien. Nous avons ensuite purifié l’enzyme afin de caractériser ses fonctions biologiques. En étudiant l’activité enzymatique de cette MTase du virus Ébola sur des ARN substrats synthétiques mimant l’extrémité 5’ des ARN viraux, nous avons démontré son implication dans le processus de méthylation de la coiffe [9]. Nos observations indiquent que la méthylation de la coiffe, chez cette famille de virus, suit un ordre séquentiel obligatoire : la 2’O-méthylation du premier nucléotide en 5’ de l’ARN précède la N7-méthylation du groupement guanosine de la coiffe. Nous avons de plus observé que la MTase du virus Ébola possède de façon surprenante, en plus de son activité de méthylation de la coiffe, une activité MTase cap-indépendante très significative : cette enzyme méthyle également des ARN non coiffés sur des résidus internes à leur séquence. En utilisant des ARN substrats homo-polymériques (poly-A, poly-U, poly-A et poly-G), nous avons démontré que les méthylations concernent uniquement les résidus adénosines, et des analyses par chromatographie liquide de haute pression (HPLC) couplée à la spectrométrie de masse ont permis de démontrer que les méthylations modifient le groupement 2’OH du ribose des adénosines.
Ce travail relate, pour la première fois, l’existence de modifications épitranscriptomiques transduites par le domaine MTase de la protéine L d’un mononégavirus. Une activité de méthylation interne similaire a récemment été caractérisée chez les Flavivirus comme le virus de la Dengue et le virus Zika [10]. De récentes études démontrent également l’existence de méthylations internes au sein de la séquence des ARN génomiques de différentes familles de virus [11]. Ces méthylations sont maintenant avérées, mais leur rôle dans la réplication virale et l’échappement au contrôle cellulaire via les mécanismes de l’immunité innée restent à déterminer. Ces modifications des ARN pourraient être impliquées dans la pathogenèse du virus Ébola liée, entre autres, à sa capacité d’échapper aux réponses antivirales précoces. Ces résultats ouvrent donc de nouvelles opportunités de recherche pour mieux comprendre les fonctions des modifications épitranscriptomiques des ARN viraux dans la relation hôte/pathogène et développer des stratégies antivirales efficaces contre le virus Ébola.
| Figure 2. Modification des ARN viraux. Les différentes modifications épitranscriptomiques des ARNs viraux induites par le domaine méthyltransférase (MTase) du virus Ébola sont représentées, à savoir des méthylations de la structure coiffe en position N7 et 2’OH du premier nucléotide ainsi que les méthylations internes des adénosines des ARN viraux. La figure indique également les rôles possibles de ces méthylations dans la traduction des ARN messagers viraux et dans la régulation de la réponse antivirale. Des molécules thérapeutiques ciblant l’activité MTase du virus Ébola devraient bloquer la synthèse des protéines virales et restaurer l’immunité antivirale protectrice. |