| dc.contributor.author | Huchon, Dorothée | - |
| dc.contributor.author | Nikaido, Masato | - |
| dc.contributor.author | Okada, Norihiro | - |
| dc.date.accessioned | 2014-02-14T14:13:29Z | |
| dc.date.available | 2014-02-14T14:13:29Z | |
| dc.date.issued | 2002 | fr_FR |
| dc.identifier.citation | Huchon, Dorothée ; Nikaido, Masato ; Okada, Norihiro ; Éléments mobiles SINE en phylogénie, Med Sci (Paris), 2002, Vol. 18, N° 12; p. 1276-1281 ; DOI : 10.1051/medsci/200218121276 | fr_FR |
| dc.identifier.issn | 1958-5381 | fr_FR |
| dc.identifier.uri | http://hdl.handle.net/10608/5106 | |
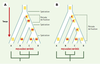
| dc.description.abstract | Les SINE (short interspersed repetitive elements) sont des éléments mobiles de l’ADN dérivés principalement des ARN de transfert ou de l’ARN cytoplasmique 7SL. Ils forment une composante majeure du génome des eucaryotes, puisque leur nombre peut atteindre plus de 104 copies par génome. Aucune fonction évidente n’est encore aujourd’hui attribuée à ces éléments. Récemment, ces « ADN égoïstes » se sont révélés être des outils très efficaces en systématique moléculaire: en effet, l’insertion d’un SINE à un site donné est un événement unique et non réversible à l’échelle des génomes. Les SINE se révèlent ainsi des outils phylogénétiques « parfaits ». L’étude de la présence ou de l’absence de sites d’insertion de ces éléments transposables a récemment conduit à des résultats phylogénétiques inattendus. Les qualités et les limites de ce nouvel outil phylogénétique sont présentées dans cet article. | fr |
| dc.description.abstract | SINE (short interspersed repetitive elements) are retroposons derived from tRNA or 7SL RNA. These repetitive sequences represent a large part of the eukaryotic genome. Their copy number can reach more than 104 per genome. However, no evident function is recognized to these elements. Recently, these « selfish DNAs » appear as a powerful tool in molecular systematic. SINE retroposons have the ability to duplicate and to be reincorporated many times into the genome. Their insertions have two major characteristics: first, a SINE has a negligible probability to be inserted twice at a specific genomic location, second, the chance that a deletion at one insertion site matches exactly the boundaries of the SINE is also insignificant. For these reasons, the analysis of SINE insertions is a source of phylogenetic information, free of convergence and reversal. SINEs thus appear as « perfect » phylogenetic characters and the study of their insertion site has recently led to unexpected phylogenetic results. The advantages and limits of this new phylogenetic method are presented here. | en |
| dc.language.iso | fr | fr_FR |
| dc.publisher | EDK | fr_FR |
| dc.relation.ispartof | M/S Revues : Articles de Synthèse | fr_FR |
| dc.rights | Article en libre accès | fr |
| dc.rights | Médecine/Sciences - Inserm - SRMS | fr |
| dc.source | M/S. Médecine sciences [ISSN papier : 0767-0974 ; ISSN numérique : 1958-5381], 2002, Vol. 18, N° 12; p. 1276-1281 | fr_FR |
| dc.title | Éléments mobiles SINE en phylogénie | fr |
| dc.type | Article | fr_FR |
| dc.contributor.affiliation | Tokyo Institute of Technology, Faculty of Bioscience and Biotechnology, 4259 Nagatsuta-cho, Midori-ku, Yokohama 226-8501, Japon | fr_FR |
| dc.identifier.doi | 10.1051/medsci/200218121276 | fr_FR |