Cartographie à haute résolution par hybridation in situ fluorescente sur fibres d'ADN décondensées
Date
1997Auteur
Monier, K
Hazzouri, M
Mongelard, F
Rousseaux, S
Vourc'h, C
Robert-Nicoud, M
Voir/
Metadata
Afficher la notice complèteRésumé
Récemment, plusieurs techniques
de préparation de fibres d’ADN décondensées sur des
lames de verre ont été décrites dans la littérature Ces
méthodes, couplées à l’hybridation in situ fluorescente,
permettent de réaliser de la cartographie des génomes
à haute résolution sur des fibres d’ADN génomique
obtenues, soit directement à partir de cellules traitées,
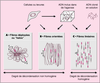
soit à partir d’ADN cloné. Trois types de fibres d’ADN
peuvent être obtenus : (1) fibres déployées
ou « halos », obtenues à partir de cellules entières,
restant attachées à une structure nucléaire résiduelle ;
(2) fibres orientées ; et (3) fibres linéaires ou ADN
« peigné », obtenues à partir d’ADN purifié en solution ou
d’ADN de haut poids moléculaire préparés
dans des blocs d’agarose. Seule la technique du peignage
moléculaire permet la conversion instantanée
des distances mesurées en microns (μm)
en une longueur en kilobases (kb) car le degré
de décondensation des fibres est reproductible.
Un facteur de conversion de 0,5 μm/kb peut être
appliqué à toutes les types de molécules d’ADN
d’une taille comprise entre 1 et 500 kb. Several techniques for preparing decondensed DNA fibers on glass surfaces have recently been described in the litterature. These techniques allow high resolution genome mapping by fluorescent in situ hybridisation on genomic DNA fibers obtained either directly from lysed cells or from cloned DNA. Using these methods, three types of DNA fibers can be obtained: 1) DNA loops or 'halo-like' fibers, 2) oriented fibers and 3) linear fibers, also known as 'combined' DNA. In the first category, DNA fibers are obtained from whole cells and are anchored to a residual nuclear structure. Linear or oriented fibers are obtained by using purified DNA in solution or high molecular weight DNA prepared in agarose blocks. For each of these methods, the degree and reproducibility of DNA stretching as well as the linearity of the resulting fibers are assessed. The advantages and drawbacks of each technique are discussed according to its efficiency for genome mapping and genomic rearrangement analysis, especially for the study of small size regions. The direct conversion of distance measurements from microns (mum) into kilobases (kb) is only possible if the degree of stretching of the DNA fiber is reproducible. In this respect molecular combing is more reliable than the other methods, since a reproducible conversion factor of 0.5 mum/kb can be applied to any DNA molecule in the range of 1 kb to 500 kb. [References: 15]
Pour citer ce document
Monier, K ; Hazzouri, M ; Mongelard, F ; Rousseaux, S ; Vourc'h, C ; Robert-Nicoud, M, Cartographie à haute résolution par hybridation in situ fluorescente, Med Sci (Paris), 1997, Vol. 13, N° 11; p.1306-11



