| dc.contributor.author | Fisette, Jean-François | - |
| dc.contributor.author | Michelle, Laetitia | - |
| dc.contributor.author | Revil, Timothée | - |
| dc.contributor.author | Chabot, Benoit | - |
| dc.date.accessioned | 2015-04-29T07:32:44Z | |
| dc.date.available | 2015-04-29T07:32:44Z | |
| dc.date.issued | 2009 | fr_FR |
| dc.identifier.citation | Fisette, Jean-François ; Michelle, Laetitia ; Revil, Timothée ; Chabot, Benoit ; Guider et intégrer pour un épissage diversifié, Med Sci (Paris), 2009, Vol. 25, N° 2; p. 175-180 ; DOI : 10.1051/medsci/2009252175 | fr_FR |
| dc.identifier.issn | 1958-5381 | fr_FR |
| dc.identifier.uri | http://hdl.handle.net/10608/6581 | |
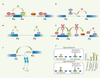
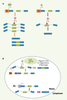
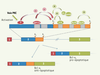
| dc.description.abstract | Trente années se sont écoulées depuis la découverte de l’épissage alternatif et notre compréhension des mécanismes de régulation demeure encore très incomplète malgré l’importance de ce processus dans les maladies humaines. Des percées récentes ont néanmoins permis d’identifier une panoplie d’activateurs et de répresseurs liant des séquences introniques et exoniques. L’expression différentielle de ces facteurs et leur utilisation combinée permettraient à la cellule d’effectuer le choix des sites d’épissage de façon précise et spécifique. Afin de conjuguer ces décisions avec d’autres processus cellulaires, certains de ces régulateurs sont recrutés durant la transcription, et leur activité est souvent intégrée aux voies de signalisation. Des efforts soutenus combinant approches classiques et technologies de pointe devraient confirmer le rôle exceptionnel de l’épissage alternatif comme agent diversificateur du protéome et améliorer notre connaissance des réseaux de régulation sous-jacents. | fr |
| dc.description.abstract | Recent studies directed at understanding alternative splicing control have produced an expanding list of regulators that can enhance or silence the use of splice sites by binding to specific sequences. A fine balance in the expression and the combinatorial use of these factors would help to adapt splicing decisions to a variety of situations. Additional levels of control are provided by tightly connecting the activity of alternative splicing factors with other cellular processes such as signal transduction and transcription. Combining classical experiments and high-throughput approaches is now confirming the important contribution of alternative splicing to proteomic diversity while helping to decipher the underlying networks of splicing regulation. | en |
| dc.language.iso | fr | fr_FR |
| dc.publisher | EDK | fr_FR |
| dc.relation.ispartof | M/S revues : Synthèse | fr_FR |
| dc.rights | Article en libre accès | fr |
| dc.rights | Médecine/Sciences - Inserm - SRMS | fr |
| dc.source | M/S. Médecine sciences [ISSN papier : 0767-0974 ; ISSN numérique : 1958-5381], 2009, Vol. 25, N° 2; p. 175-180 | fr_FR |
| dc.subject.mesh | Épissage alternatif | fr |
| dc.subject.mesh | Animaux | fr |
| dc.subject.mesh | Régulation de l'expression des gènes | fr |
| dc.subject.mesh | Variation génétique | fr |
| dc.subject.mesh | Homéostasie | fr |
| dc.subject.mesh | Humains | fr |
| dc.subject.mesh | Modèles génétiques | fr |
| dc.subject.mesh | Régions promotrices (génétique) | fr |
| dc.subject.mesh | Protéome | fr |
| dc.subject.mesh | Précurseurs d'ARN | fr |
| dc.subject.mesh | ARN messager | fr |
| dc.subject.mesh | Transcription génétique | fr |
| dc.title | Guider et intégrer pour un épissage diversifié | fr |
| dc.title.alternative | Guiding and integrating to control and diversify splicing | en |
| dc.type | Article | fr_FR |
| dc.contributor.affiliation | Chaire de recherche du Canada en génomique fonctionnelle, Groupe ARN, Département de microbiologie et d’infectiologie, Faculté de médecine et des sciences de la santé, Université de Sherbrooke, 3001, 12e Avenue Nord, Sherbrooke, Québec J1H 5N4, Canada | fr_FR |
| dc.identifier.doi | 10.1051/medsci/2009252175 | fr_FR |
| dc.identifier.pmid | 19239850 | fr_FR |