| dc.contributor.author | Mignotte, B | fr_FR |
| dc.contributor.author | Zamzami, N | fr_FR |
| dc.contributor.author | Petit, P | fr_FR |
| dc.contributor.author | Vayssière, JL | fr_FR |
| dc.contributor.author | Kroemer, G | fr_FR |
| dc.date.accessioned | 2012-08-23T07:49:21Z | |
| dc.date.available | 2012-08-23T07:49:21Z | |
| dc.date.issued | 1998 | fr_FR |
| dc.identifier.citation | Mignotte, B ; Zamzami, N ; Petit, P ; Vayssière, JL ; Kroemer, G, Contrôle mitochondrial de l'apoptose : la mort cellulaire programmée est-elle apparue à la suite de l'événement endosymbiotique à l'origine des mitochondries ?, Med Sci (Paris), 1998, Vol. 14, N° 1; p.54-60 | fr_FR |
| dc.identifier.issn | 1958-5381 | fr_FR |
| dc.identifier.uri | http://hdl.handle.net/10608/883 | |
| dc.description.abstract | La plupart des cellules animales peuvent mourir par un
processus de mort cellulaire que l’on appelle apoptose.
Les protéines cellulaires impliquées dans ce processus
d’autodestruction ont relativement peu changé
depuis la dichotomie nématodes/vertébrés. Il apparaît
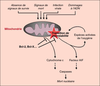
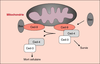
maintenant que les mitochondries occupent une place
déterminante dans le contrôle de l’apoptose. Chez les
mammifères, comme chez les nématodes, les protéines de la
famille Bcl-2/Bcl-X/Ced-9 inhibent l’apoptose au moins
à deux niveaux : régulation du passage d’ions ou de
molécules pro-apoptotiques au travers de pores
transmembranaires, et ancrage, au niveau des
mitochondries, de protéines impliquées dans la
transduction de signaux apoptotiques.
Par ailleurs, il est maintenant admis que les
mitochondries sont des endosymbiontes ayant pour origine
une bactérie aérobie qui aurait été absorbée par l’ancêtre
des cellules eucaryotes. Une partie de la machinerie
apoptotique existerait chez des eucaryotes unicellulaires et
certains des composants contrôlant l’apoptose pourraient
même être présents chez les bactéries. Il est donc possible
que le mécanisme à l’origine du maintien de la symbiose
entre la bactérie ancêtre des mitochondries et la cellule
hôte à l’origine des eucaryotes ait fourni les bases d’un
contrôle de la survie cellulaire. Les métazoaires auraient
exploité cette possibilité en connectant les effecteurs
mitochondriaux de la mort cellulaire aux voies de
transduction de signaux. | fr |
| dc.description.abstract | Most animal cells can undergo a process of programmed cell death called apoptosis. Cellular proteins involved in this self-destruction process have changed relatively little since the divergence of nematodes and vertebrates. It appears now that mitochondria occupy a strategic place in the control of apoptosis. In mammals, as well as in nematodes, proteins of the Bcl-2/Bcl-X/Ced-9 family inhibit apoptosis at least at two levels: regulation of membrane permeability to ions or pro-apoptotic molecules, and anchorage to the mitochondria of proteins involved in the transduction of apoptotic signals. It is now accepted that mitochondria are endosymbionts, originating in aerobic bacteria which were integrated by the ancestor of eukaryotic cells. A part of the apoptotic machinery could exist in unicellular eukaryotes and some components controlling apoptosis might be present in prokaryotes. It is therefore possible that the mechanism originally involved in the maintenance of the symbiosis between the bacterial ancestor of the mitochondria and the host cell precursor of eukaryotes, provided the basis for the actual mechanism controlling cell survival. Metazoans would have exploited this possibility by connecting the mitochondrial effecters of cellular death to the pathways of signal transduction. [References: 46] | en |
| dc.language.iso | fr | fr_FR |
| dc.publisher | Masson, Paris | fr_FR |
| dc.rights | Article en libre accès | fr |
| dc.rights | Médecine/Sciences - Inserm - SRMS | fr |
| dc.source | M/S. Médecine sciences [revue papier, ISSN : 0767-0974], 1998, Vol. 14, N° 1; p.54-60 | fr_FR |
| dc.title | Contrôle mitochondrial de l'apoptose : la mort cellulaire programmée est-elle apparue à la suite de l'événement endosymbiotique à l'origine des mitochondries ? | fr |
| dc.title.alternative | Mitochondrial control of apoptosis : did programmed cell death appear after the endosymbiotic event giving rise to mitochondria ? | fr_FR |
| dc.type | Article | fr_FR |
| dc.contributor.affiliation | Universite de Versailles-Saint-Quentin, EA1636, 91198 Gif-sur-Yvette, France; INSERM, Cnrs UPR 420, IRC, 94800 Villejuif, France; Centre de genetique moleculaire du Cnrs UPR 9061, France | - |
| dc.identifier.doi | 10.4267/10608/883 | |