| dc.contributor.author | Croteau, Félix R. | - |
| dc.contributor.author | Rousseau, Geneviève M. | - |
| dc.contributor.author | Moineau, Sylvain | - |
| dc.date.accessioned | 2019-11-05T12:46:31Z | |
| dc.date.available | 2019-11-05T12:46:31Z | |
| dc.date.issued | 2018 | |
| dc.identifier.citation | Croteau, Félix R. ; Rousseau, Geneviève M. ; Moineau, Sylvain ; Le système CRISPR-Cas : Au-delà de l’édition génomique, Med Sci (Paris), , Vol. 34, N° 10 ; p. 813-819 ; DOI : 10.1051/medsci/2018215 | |
| dc.identifier.issn | 1958-5381 | |
| dc.identifier.uri | http://hdl.handle.net/10608/9870 | |
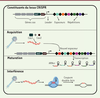
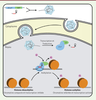
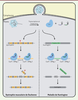
| dc.description.abstract | CRISPR-Cas est un système immunitaire adaptatif utilisé par de nombreux microbes pour se défendre contre l’invasion d’acides nucléiques tels que les génomes viraux et autres éléments génétiques mobiles. Le système microbien utilise son locus CRISPR pour stocker de l’information génétique afin de produire des ARN guides. Ces derniers, de concert avec des endonucléases (Cas), empêchent des invasions futures. Des parties de ce système microbien ont été exploitées pour développer un puissant outil d’édition des génomes dans une panoplie d’organismes. La capacité de CRISPR-Cas9 à couper efficacement et à des endroits très précis de l’ADN pourrait peut-être permettre un jour de guérir certaines maladies génétiques humaines. La malléabilité de cet outil d’édition rend possible une variété d’applications allant de la modulation de l’expression de gènes à des modifications épigénétiques. Les locus CRISPR représentent également une mine d’informations pouvant servir de méthode de typage de souches microbiennes ou encore une façon d’étudier les interactions entre les bactéries et leurs habitats. | fr |
| dc.description.abstract | CRISPR-Cas is an adaptive immune system used by many microbes to defend against nucleic acids invasion such as viral genomes. The microbial system uses its CRISPR locus to store genetic information that will generate short CRISPR RNAs. The latter with endonucleases (Cas) prevent future viral infections. Parts of this system were exploited to develop a powerful genome editing tool that was adapted for a variety of organisms. The ability of the CRISPR-Cas9 technology to effectively and precisely cut a targeted genomic DNA region has the potential to may be one day cure genetic diseases. The malleability of this editing tool also offers a wide range of possibilities from modulations of gene expression to epigenetic modifications. The natural CRISPR loci found in bacteria can be used to differentiate microbial strains or to study the interactions between bacteria and its habitat. Addressing CRISPR-Cas fundamentals in microbes and its popular use in eukaryotes, this review presents an update on a system that has revolutionized biological sciences. | en |
| dc.language.iso | fr | |
| dc.publisher | EDP Sciences | |
| dc.relation.ispartof | M/S Revues | |
| dc.rights | Article en libre accès | fr |
| dc.rights | Médecine/Sciences - Inserm - SRMS | fr |
| dc.source | M/S. Médecine sciences [ISSN papier : 0767-0974 ; ISSN numérique : 1958-5381], , Vol. 34, N° 10; p. 813-819 | |
| dc.subject.mesh | Animaux | fr |
| dc.subject.mesh | Systèmes CRISPR-Cas | fr |
| dc.subject.mesh | Édition de gène | fr |
| dc.subject.mesh | Génie génétique | fr |
| dc.subject.mesh | Thérapie génétique | fr |
| dc.subject.mesh | Génome | fr |
| dc.subject.mesh | Humains | fr |
| dc.subject.mesh | physiologie | fr |
| dc.subject.mesh | méthodes | fr |
| dc.subject.mesh | tendances | fr |
| dc.title | Le système CRISPR-Cas : Au-delà de l’édition génomique | fr |
| dc.title.alternative | The CRISPR-Cas system: beyond genome editing | en |
| dc.type | Article | |
| dc.contributor.affiliation | Département de biochimie, de microbiologie, et de bio-informatique, faculté des sciences et de génie, groupe de recherche en écologie buccale, faculté de médecine dentaire, Université Laval, Québec, QC, G1V 0A6, Canada | |
| dc.contributor.affiliation | Félix d’Hérelle reference center for bacterial viruses, faculté de médecine dentaire, Université Laval, 1045 avenue de la Médecine, Québec, QC, G1V 0A6, Canada | |
| dc.identifier.doi | 10.1051/medsci/2018215 | |
| dc.identifier.pmid | 30451675 | |